作者:xiaolanLin
声明 :本文版权归作者和博客园共有,来源网址:https://www.cnblogs.com/xiaolan-Lin
一个不是学生物的孩子来搞生物,当真是变成了一块废铁啊,但也是让我体会到了一把生物信息的力量。
废话不多说,开整!
任务:快速高效从PubMed上下载满足条件的文献PMID、标题(TI)、摘要(AB)。
PubMed官网 https://pubmed.ncbi.nlm.nih.gov
此处有几种选择可以达到目的:
(1)官网上匹配筛选条件(注:匹配快速,但是下载下来的数量受到限制,每次只能下载10000条数据,甚至更少。)
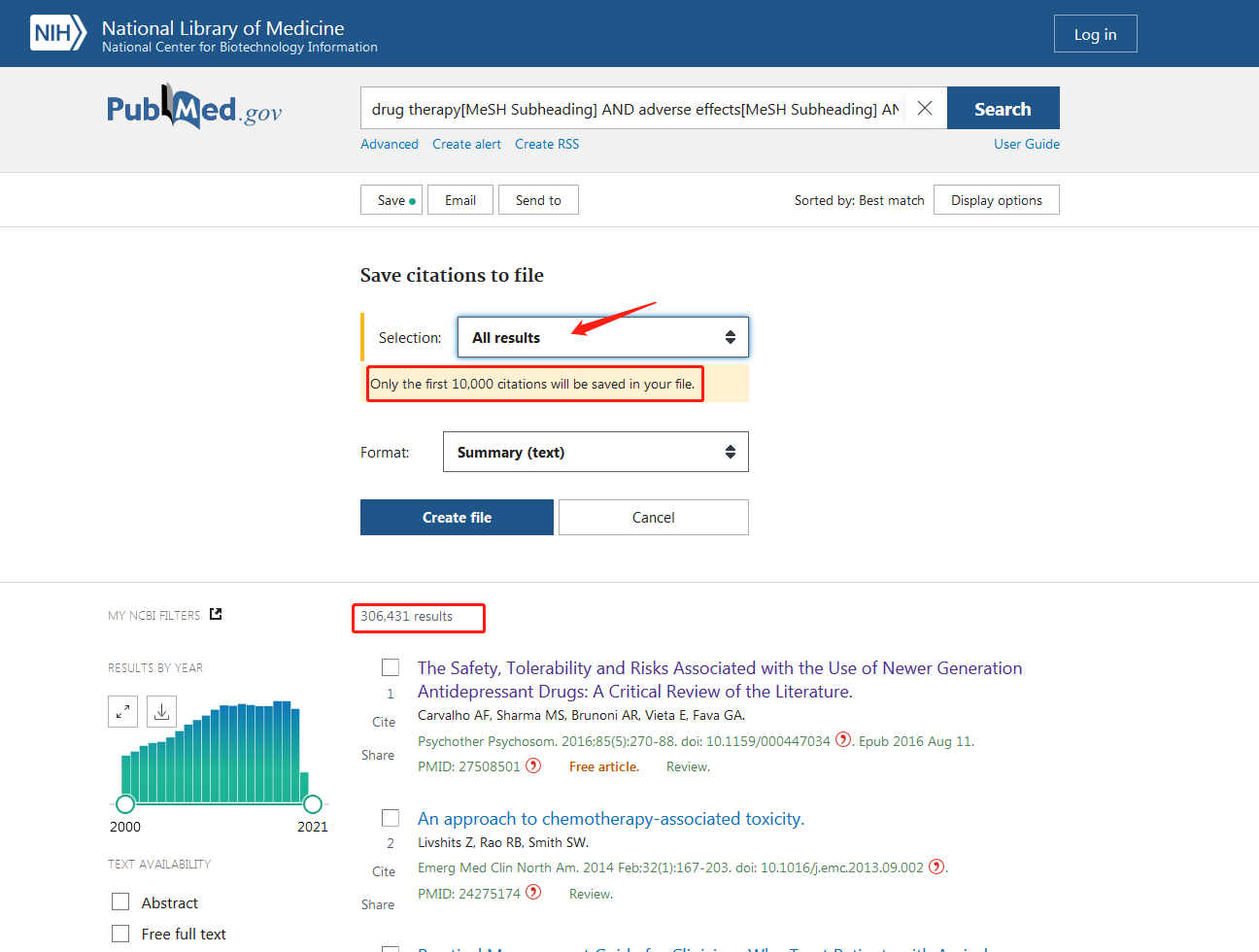
可以看到,我需要的数据是有三十多万条,但是每次只能下载10000条,那我岂不是要手动n次。。很明显,在大批量下载文献的情况下,官网不是很友好。
(2)R语言有个R包,叫做easyPubMed,这里我也给大家贴上学习指南(https://cran.r-project.org/web/packages/easyPubMed/vignettes/getting_started_with_easyPubMed.html)
由于我不喜欢用R写代码,所以我写一半还是换了Python,熟练R的小伙伴可以自行根据指南走通需求。
(3)重量级库来了,Python自带的Bio包中的Entrez检索库,简直就是我的救星,以下是我的代码:
注:Entrez在Bio包中,Bio的安装请移步 https://www.cnblogs.com/xiaolan-Lin/p/14023147.html
import numpy as np
from Bio import Medline, Entrez # 一般是通过BioPython的Bio.Entrez模块访问Entrez
from collections import Counter
Entrez.email = "(此处写你自己在官网注册的邮箱账号)" # 应用自己的账号访问NCBI数据库
# 此处需将服务器协议指定为1.0,否则会出现报错。http.client.IncompleteRead: IncompleteRead(0 bytes read)
# 服务器http协议1.0,而python的是1.1,解决办法就是指定客户端http协议版本
import http.client
http.client.HTTPConnection._http_vsn = 10
http.client.HTTPConnection._http_vsn_str = 'HTTP/1.0'
"""
Entrez 是一个检索系统,可以用其访问NCBI数据库,比如说PubMed,GenBank,GEO等。
获得有关 global PBDE 的所有文献的PubMed IDs
"""
# handle_0 = Entrez.esearch(db="pubmed", term="drug therapy[Subheading] AND adverse effects[Subheading] AND humans[MeSH Terms]", retmax=306431)
handle_0 = Entrez.esearch(db="pubmed", term="drug therapy[MeSH Subheading] AND adverse effects[MeSH Subheading] AND humans[MeSH Terms] AND (2000/01/01[Date - Publication] : 2021/12/31[Date - Publication])",
ptyp="Review", usehistory="y", retmax=306431)
record = Entrez.read(handle_0) # 获取检索条件的所有文献
idlist = record["IdList"] # 提取出文献id
print ("Total: ", record["Count"])
No_Papers = len(idlist) # 共306431篇文献 2000-01-01:2021-12-31
webenv = record['WebEnv']
query_key = record['QueryKey']
total = No_Papers
step = 1300
print("Result items:", total)
with open("./Data_PubMed/PBDE1.txt", 'w') as f:
for start in range(0, total, step):
print("Download record %i to %i" % (start + 1, int(start + step)))
handle_1 = Entrez.efetch(db="pubmed", retstart=start, rettype="medline", retmode="text",
retmax=step, webenv=webenv, query_key=query_key) # 获取上述所有文献的PubMed IDs
records = Medline.parse(handle_1)
records = list(records) # 将迭代器转换至列表(list)
for index in np.arange(len(records)):
id = records[index].get("PMID", "?")
title = records[index].get("TI", "?")
title = title.replace('[', '').replace('].', '') # 若提取的标题出现[].符号,则去除
abstract = records[index].get("AB", "?")
f.write(id)
f.write("\n")
f.write(title)
f.write("\n")
f.write(abstract)
f.write("\n")
话不多说,结果跑出来了我真的很快乐~

最后的结果是存放在txt文件中,大伙儿根据自己的需求改变代码所需字段啊。
现在我来解释一下,我贴上的这串代码的实现原理,首先是通过Entrez检索到符合我筛选条件的文献,里边的限制条件包括了几个词汇匹配以及时间限制,时间我限制在了2000年1月1日到2021年的12月31日(这里的时间我选用的是Date - Publication,时间选取Date - Completion、Date - Modification还是Date - Publication其实还是有争议的,大家自行考虑选取)。
Entrez.esearch的作用就是用来检索的,里边的参数db指向你要检索的数据库,代码中的注释也写了,Entrez作为一个接口检索,除了能够检索PubMed中的文献,也能去到别的数据库检索文献;term是写你的筛选语句,注意你写的检索语句不能带有引号,单引号也不行,否则会检索不到,如果不知道检索语句怎么写,或者是不知道字段是否被定义,可以在官网的检索那里https://pubmed.ncbi.nlm.nih.gov/advanced/选择字段输入内容自动生成query,但是生成的语句是不太智能的,会有很多括号是你不需要的,自己写代码的时候要适当去掉;ptyp我这里用的是Review,usehistory是y,意思是后边我的检索要记住这个语句,根据历史查询来检索;retmax如果不进行设置的话,默认给你的最大数据量好像是只有1000,我要的检索内容是超过这个值的,因此我需要自定义检索的数量。
Entrez.read是对Entrez.esearch检索到的内容进行读取,里边包含了9种元素,我们主要是想从中得到文献的id号,只有拿到了文献的id号,我们后面进行摘要的提取才能准确定位。
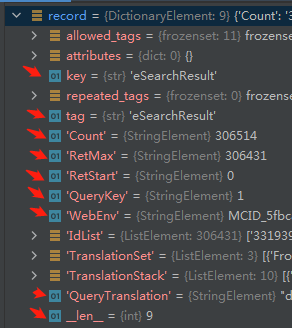
最后是循环当中步长的设置,这里就要根据自己的需求来定义了,包括内容的提取,因为我只需要PMID、标题(TI)、摘要(AB),所以我就没有加载别的内容进来,这里也有一点要注意,标题下载下来是大部分带有[ ].的,方便操作我直接就在下载的时候给去除了,这也是上面replace代码的由来。
附上我参考的链接,如果我这篇文章解决不了你的问题,那么希望下面的渠道能够帮助到你
https://zhuanlan.zhihu.com/p/54611852
https://zhuanlan.zhihu.com/p/262957260
以上就是Python 利用Entrez库筛选下载PubMed文献摘要的示例的详细内容,更多关于Python Entrez库下载PubMed文献的资料请关注易盾网络其它相关文章!
