在遗传学中,当两个基因相互作用然后导致对应性状的出现,说明两个基因间存在相互作用。在之前的文章中,介绍了很多的基因相互作用模型,列表如下 互补作用 积加作用 重复作
在遗传学中,当两个基因相互作用然后导致对应性状的出现,说明两个基因间存在相互作用。在之前的文章中,介绍了很多的基因相互作用模型,列表如下
其中上位作用研究的作为广泛,作为GWAS研究中使用的最广泛的工具,plink当然也支持上位作用,即Epistasis的分析,有以下两种方式
1. fast-epistasis
这种用法适合case/control 二分类的表型,原理和Allele水平的GWAS分析一样,基本用法如下
plink \--bfile input \
--fast-epistasis \
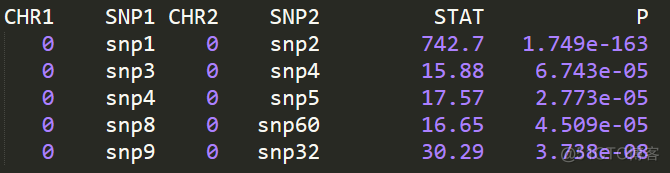
输出结果示意如下
2. epistasis
这种用法适用范围广,case/control还有连续表型都适用,通过线性回归或者逻辑回归来进行分析,基本用法如下
plink \--bfile input \
--epistasis \
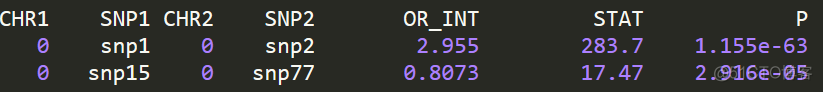
输出结果示意如下
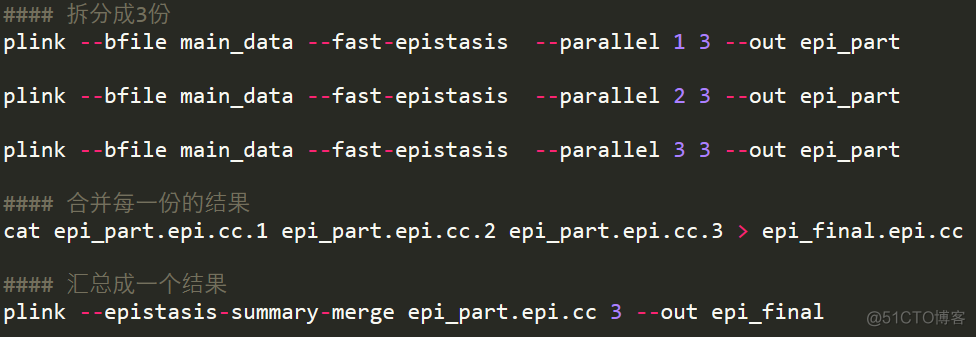
当SNP位点的数量很多时,两两相互作用分析的计算量是很大的,为了加速运行,plink支持拆分后并行,用法如下
当然,以上只是基本用法,还是有很多的参数可以调整,更多的细节请参考官方文档。
·end·

生物信息入门
只差这一个
公众号
