在生物信息数据分析中,R语言是必备技能,简洁的语法,丰富的生态,美观的可视化,种种优势使得其成为该领域中使用最广泛的编程语言之一。用R进行meta分析当然也是可以的,本文要介绍的R包rmeta, 就是其中之一,可以用于gwas meta分析,支持随机效应模型和固定效应模型,官方文档如下
https://cran.r-project.org/web/packages/rmeta/rmeta.pdf
作为CRAN的一员,其安装方式如下
install.packages(“rmeta”)
首先来看下其输入文件格式,其输入文件并不是常规的GWAS分析结果, 比如pvalue, beta等统计指标,而是原始的频数分布,而且是二分类数据,比如两种Allele的频数分布,对于每个study, 统计如下所示的表格数据
Allele
A
a
Case
a
b
Control
c
d
也就是说,对于每个study,我们需要有4个数值。软件内置的示例数据如下
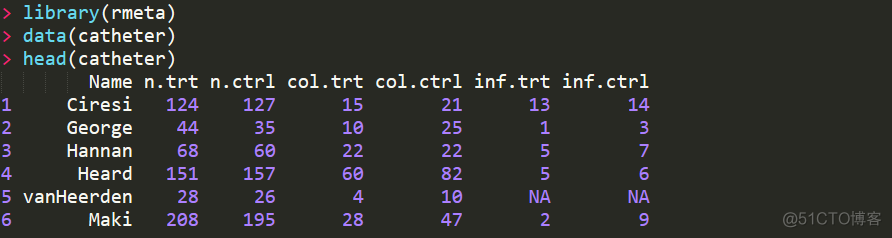
这个数据列数很多,真正进行分析时,只需要其中的前4列数据。在该R包中,两种模型对应的函数如下

以固定效应模型为例,进行meta分析的代码如下
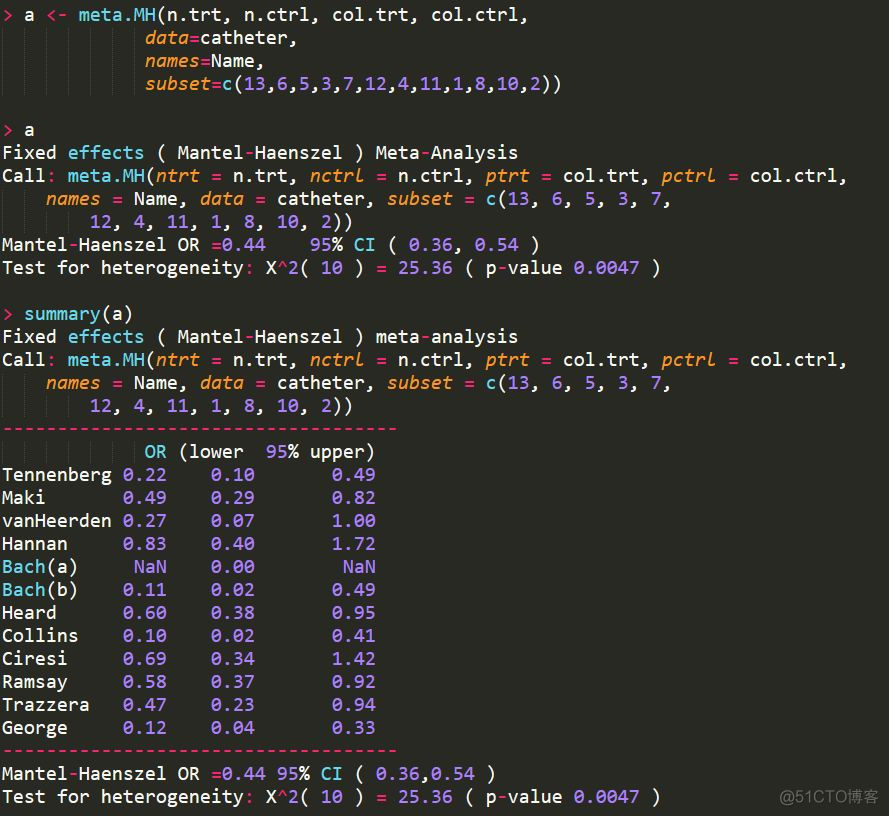
从代码可以看出,分析时只使用了前4列,对应每个study的2X2表格数据,因为部分study中有缺失值NA, 有缺失值的数据是没法分析的,所以通过subset函数选取了其中的部分行。
该R包最大的特点是其可视化功能,对于meta分析的结果,提供了以下两种可视化的函数
1. metaplot
该函数用于展示每个study的名称和对应OR值的分布,以及meta分析后最终计算出的OR值,用法如下
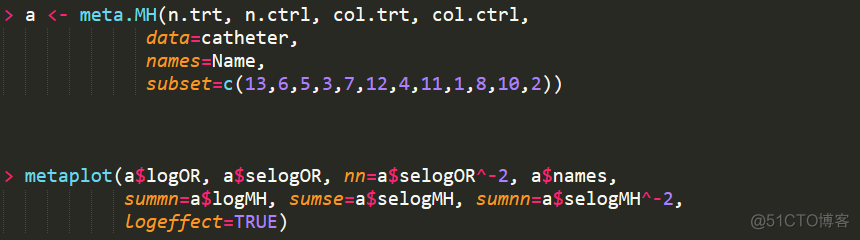
输出结果如下
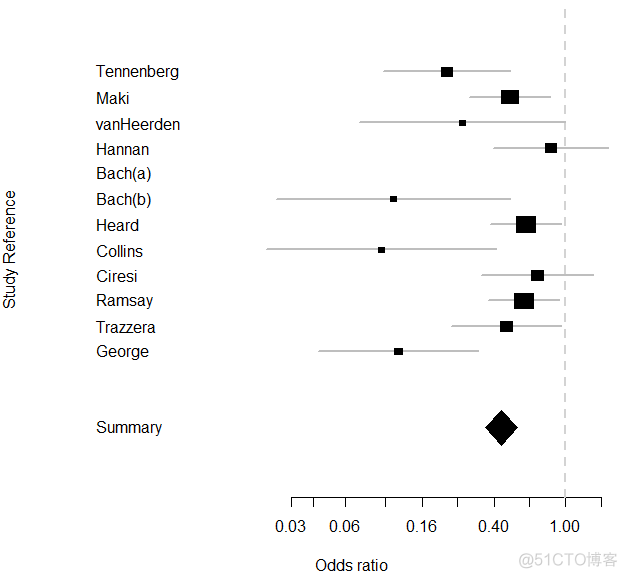
2. forestplot
该函数用于自定义的展示很多额外的文字信息,用法如下
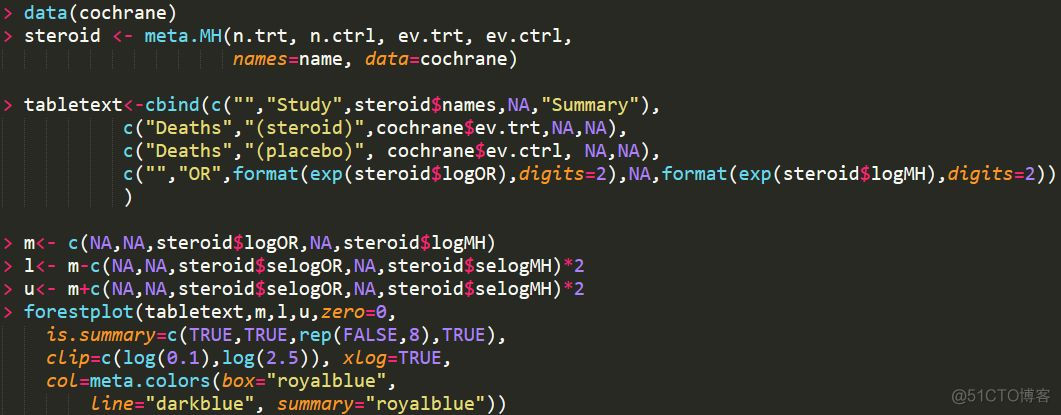
输出结果如下
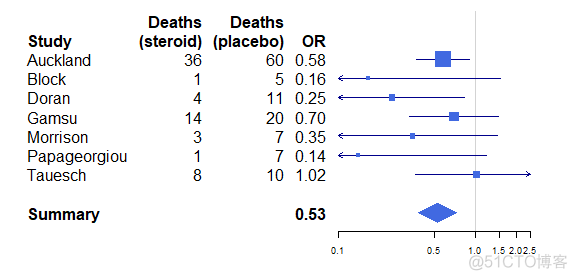
通过rmeta可以方便个性化的绘制森林图,更多用法请参考官方的帮助文档。
·end·

生物信息入门
只差这一个
公众号
