MDS是一种常用的降维算法,其基本思想是保证高维空间映射到低维空间之后,样本间的相对距离基本不变。
根据所用的相对距离的具体指标,可以划分成以下两大类
1. metric multidimensional scaling
2. non-metric multidimensional scaling, 缩写为NMDS
区别在于,metric MDS采用的是真实的距离指标,比如欧式距离,而NMDS并不是直接采用距离,而是采用距离的秩序。在数学上,距离这个概念需要满足以下4个特性
1. 非负性
2. 同一性
3. 对称性
4. 值低性
非负性指的是两个样本间的距离要大于等于零,公式如下
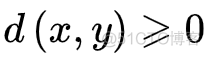
同一性表示样本和自身的距离为0,公式如下

对称性指的是两个样本相互之间的距离相等,公式如下
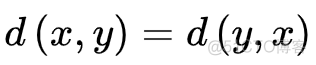
值递性指的是对于3个样本构成的三角形,任意一条边都小于等于另外两条边之和,公式如下
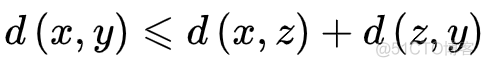
符合以上4个特性的指标才可以称之为距离,比如欧式距离就符合上述定义。除了距离以外,还有一个概念,叫做不相似度dissimilarity, 与距离相比,不相似度也具有非负性,同一性和对称性,但是不具有值递性。
以欧式距离为例,MDS要实现将原始的D维空间投影到低维空间Z, 并保持降维前后,样本点之间的距离不变,对应的公式如下
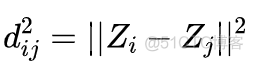
公式左侧为原始空间的样本距离,右侧为低维空间的样本距离,采用了范数的表示形式,对应l2范数,公式如下
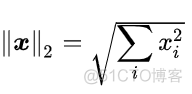
可以看到,就是一个欧式距离的求解公式。进一步,将公式右侧展开,得到以下公式
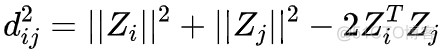
对向量Z进行标准化,令
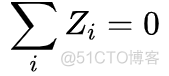
标准化的结果就是各个样本的Z向量之和为0
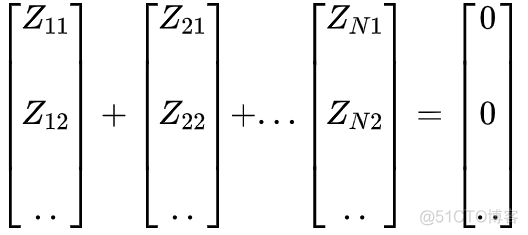
对上述的距离公式求和,可以得到如下结果
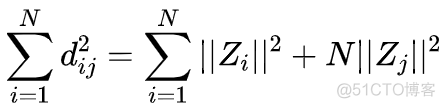
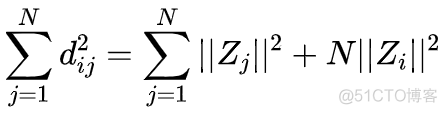
再次对上述两个公式求和,可以得到如下结果
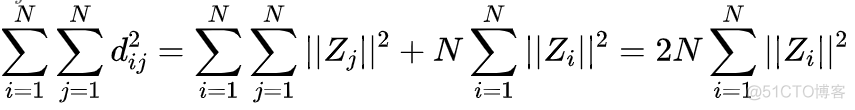
定义内积矩阵B

利用上述公式,可以推导出B的值如下
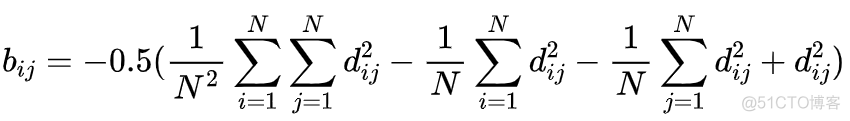
对B矩阵进行特征分解
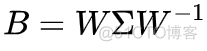
则降维之后的Z矩阵可以表示如下
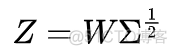
MDS算法的流程总结如下如下
1. 计算原始空间中样本点的距离矩阵
2. 计算内积矩阵B
3. 对矩阵B进行特征值分解,获得特征值矩阵和特征向量矩阵
4. 取特征值矩阵最大的前Z项及其对应的特征向量,构成最终降维之后的结果
在scikit-learn中,应用MDS降维的代码如下
>>> from sklearn.manifold import MDS>>> from sklearn.datasets import load_digits
>>> X, _ = load_digits(return_X_y=True)
>>> X.shape
(1797, 64)
>>> embedding = MDS(n_components=2)
>>> X_transformed = embedding.fit_transform(X[:100])
>>> X_transformed.shape
(100, 2)
MDS算法只需要依赖样本的距离矩阵,不需要任何其他的先验知识,降维之后保持了样本在原始空间的相对关系,可以获得很好的可视化效果。
·end·

一个只分享干货的
生信公众号
