测序数据量对于NGS数据分析是非常重要的,测序数据量过低,不能有效覆盖基因组完整信息,测序数据量过高,则会造成冗余,不够经济。为了验证当前测序量能否满足需求,或者说加大测序量是否能够进一步挖掘的更大量的信息,通常需要进行饱和度分析。
在转录组和扩增子测序中,饱和度分析应用的已经非常成熟,对于WES/WGS等基因组测序而言,对测序深度也已经基本形成了统一共识。对于ATAC而言,测序饱和度分析要怎么做呢?本片文章就来解密一下。
ATACseqQC作为一个专门针对ATAC文库进行QC的R包,在其文章中,提供了测序饱和度分析的思路和方法,文章链接如下
https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-018-4559-3
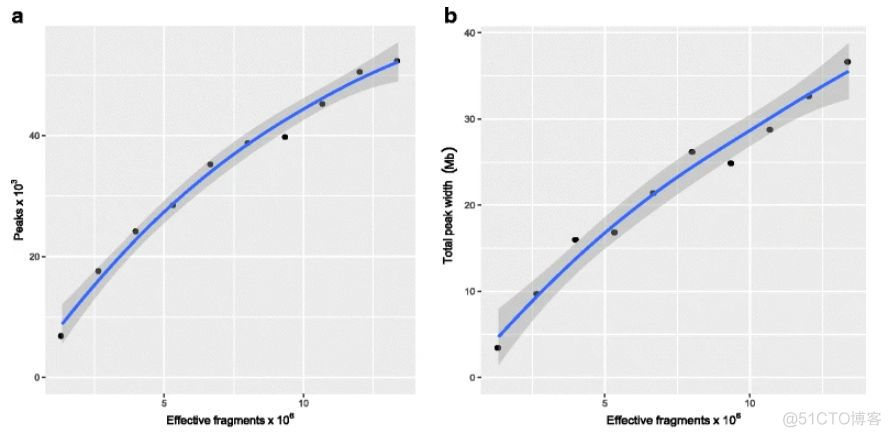
如上图所示,ATACseqQC的饱和度分析思路如下,对effectice fragment进行随机抽样,比如10%,20%到100%, 对于每个梯度分别进行peak calling, 统计其peak个数,然后以effectice fragment数目为横坐标,peak个数为纵坐标绘制散点图,并进行拟合,如果拟合曲线的末端斜率上升平缓,表明继续加大测序数据量,新识别到的peak个数也不会新增太多,就可以认为当前的测序数据量是比较饱和的,不比再加测数据量了。如果曲线末端仍处于一个斜率急剧上升的阶段,说明测序数据量不够,还需加大测序数据量。
理解了分析思路,再来看一下具体的操作过程。第一步是对effectice fragment进行抽样。所谓effectice fragment,即过滤掉PCR重复,去除了线粒体之后的有效序列。在实际分析中,质控后的clean reads首先比对到参考基因组上,生成原始的bam文件,也称为raw bam。然后对原始的bam文件进行过滤,去除MAPQ较低的alignment, 去除PCR重复,去除比对到线粒体的序列,经过重重筛选,得到了一个filter之后的bam文件,这个filter bam文件中保存的就是effectice fragment。
对effectice fragment进行抽样,本质就是操作这个bam文件。通过linux下的shuf命令对sam文件随机抽取对应的行,从而实现对effectice fragment抽样的目的,基本思路如下
在文章的附件中提供了一个bash脚本,用于对bam文件进行抽样,用法如下
bash downsample.sh GL1.bam\GL1 \0.1 0.2 0.3 0.4 0.5 \0.6 0.7 0.8 0.9
第一个参数为filter之后的bam文件,第二个参数为输出文件的前缀,其他参数为抽取的百分比,0到1之间,运行成功后,会生成一系列文件,以0.1为了,生成的文件如下
GL1.bam.name.sorted.0.1.samGL1.bam.name.sorted.0.1.coord.sorted.bam
GL1.bam.name.sorted.0.1.coord.sorted.bam.bai
利用排序好的bam文件可以直接进行peak calling, 然后统计每个抽样百分比对应的effectice fragment数目和peak个数,就可以绘制测序饱和度图了。在ATACseqQC中,提供了saturationPlot函数,可以直接读取一系列peak文件,然后绘制测序饱和度图,具体的用法可以查看该函数的帮助文档。
ATACseqQC给我们提供了一个很好的思路,来进行ATAC文库的测序饱和度分析,类似的,这个做法也可以推广到chip_seq, m6A_seq等IP类型的实验中去。
·end·

一个只分享干货的
生信公众号
