对于ATAC文库而言,其插入片段的长度分布有着非常典型的规律,示意如下 每200bp会存在一个峰,这个周期性波动反应的是核小体的个数。在ATAC_seq的数据分析中,会对插入片段长度分
对于ATAC文库而言,其插入片段的长度分布有着非常典型的规律,示意如下
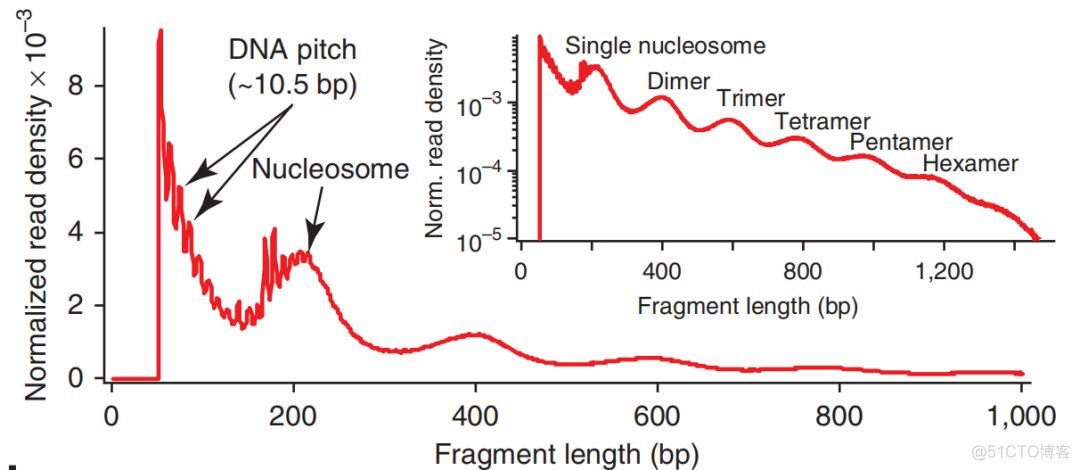
每200bp会存在一个峰,这个周期性波动反应的是核小体的个数。在ATAC_seq的数据分析中,会对插入片段长度分布进行可视化,观察其是否符合这样的周期性规律,一定程度可以反映文库构建的质量,那么如何在做这样一张分布图呢?
比对之后我们会得到bam文件,画图所需的插入片段长度就需要从bam文件中提取,需要注意,这里的插入片段是文库中adapter之间的插入片段,即fragment, 需要和insert size区别开来。
对于单端测序而言,只有bam文件是不够的,需要借助工具来预测fragment length, 这里就不展开了。对于双端测序而言,事情就变得简单了。bam文件的第9列直接存储了fragment length的信息,直接提取之后就可以用来画图,提取的代码如下
samtools view input.bam | \awk -F'\t' 'function abs(x){return ((x < 0.0) ? -x : x)} {print $1"\t"abs($9)}' | \
sort | uniq | cut -f2 > fragment.length.txt
bam文件中每一行以reads为单位,这里去重是为了避免来自同一个fragemnts的reads重复统计。提取好之后,用R画图就可以了,R代码如下
data <- read.table("fragment.length.txt", header = F)# 设置插入片段长度的阈值,过滤掉太长的片段
length_cutoff <- 1200
fragment <- data$V1[data$V1 <= length_cutoff]
# 利用直方图统计频数分布,设置柱子个数
breaks_num <- 500
res <- hist(fragment, breaks = breaks_num, plot = FALSE)
# 添加坐标原点
plot(x = c(0, res$breaks),
y = c(0, 0, res$counts) / 10^2,
type = "l", col = "red",
xlab = "Fragment length(bp)",
ylab = expression(Normalized ~ read ~ density ~ 10^2),
main = "Sample Fragment sizes")
输出结果示意如下
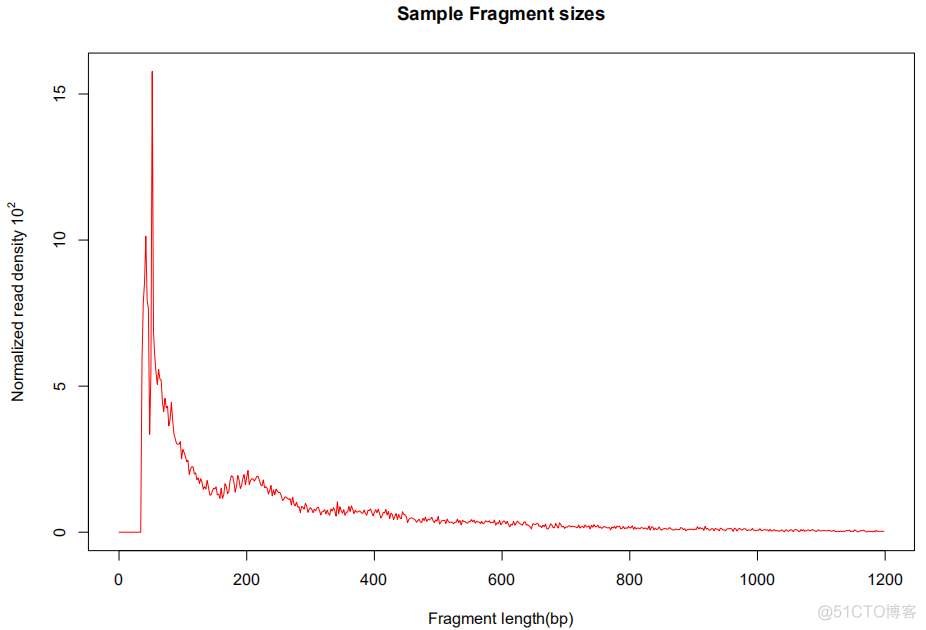
这种是最简单的方式,除此之外,还有picard的CollectInsertSizeMetrics, bedtools的bamPEFragmentSize也都可以计算插入片段长度,但是在我看来,还是本文介绍的方式最为简单直接。
·end·

一个只分享干货的
生信公众号
