maftools是一个R包,发布在bioconductor上,专门用于MAF文件中信息的可视化,链接如下
https://bioconductor.org/packages/release/bioc/vignettes/maftools/inst/doc/maftools.html
提供了非常多的可视化方案,用法也比较简便,首先读取MAF文件,代码如下
> library(maftools)> maf <- "TCGA.LAML.somatic.maf.gz"
> laml = read.maf(maf = maf)
1. summary
统计样本水平,基因水平的不同突变类型的个数,代码如下
# 样本水平> getSampleSummary(laml)
# 基因水平
> getGeneSummary(laml)
# 将summary结果输出
> write.mafSummary(maf = laml, basename = 'laml')
通过以下代码可以对summary的结果进行可视化
> plotmafSummary(maf = laml)可视化结果示意如下
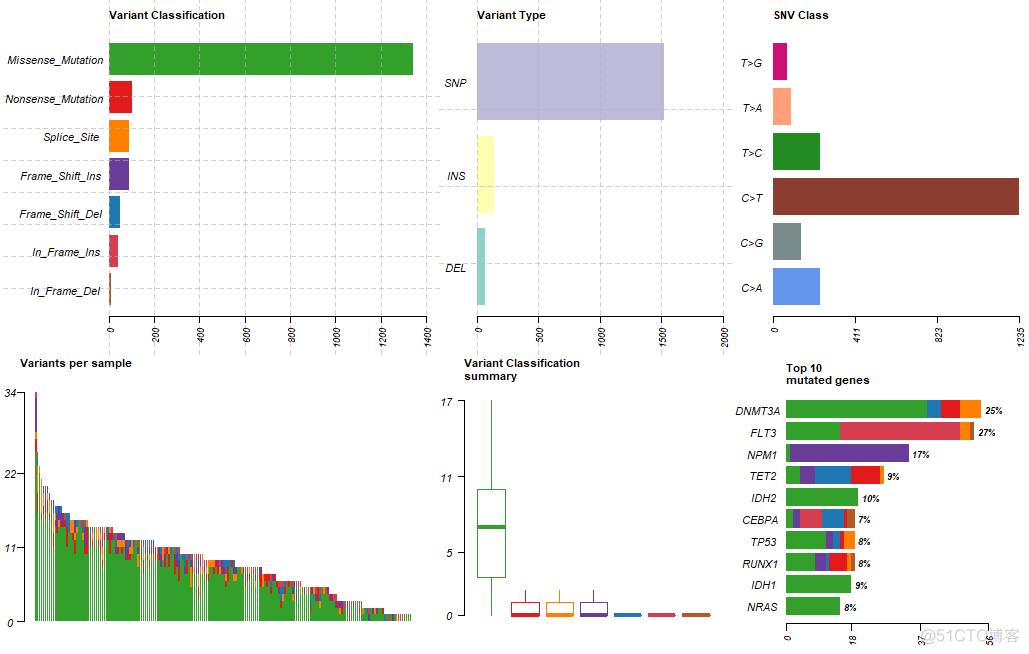
第一幅图子图是不同变异类型频数分布柱状图,根据对蛋白编码的影响划分成了Missense_Mutation等7个类型;第二幅子图是不同变异类别的频数分布柱状图,分成了SNP, INS,DEl三种类别;第三幅子图是SNV突变类型的频数分布柱状图,考虑到突变之间的组合和正负链,分成了6种类别;第四幅子图是每个样本中不同变异类型堆积柱状图,对应图一的7种类型;第五幅子图是不同变异类型在每个样本中分布的箱体图,对应图一中的7种类型,第六幅图是突变频率最高的top10个基因上的不同突变类型的堆积柱状图,对应图一的7种类型。
2. oncoplot
oncoplot是肿瘤研究中常见的可视化方式,通过以下代码可以实现
> oncoplot(maf = laml)可视化结果示意如下
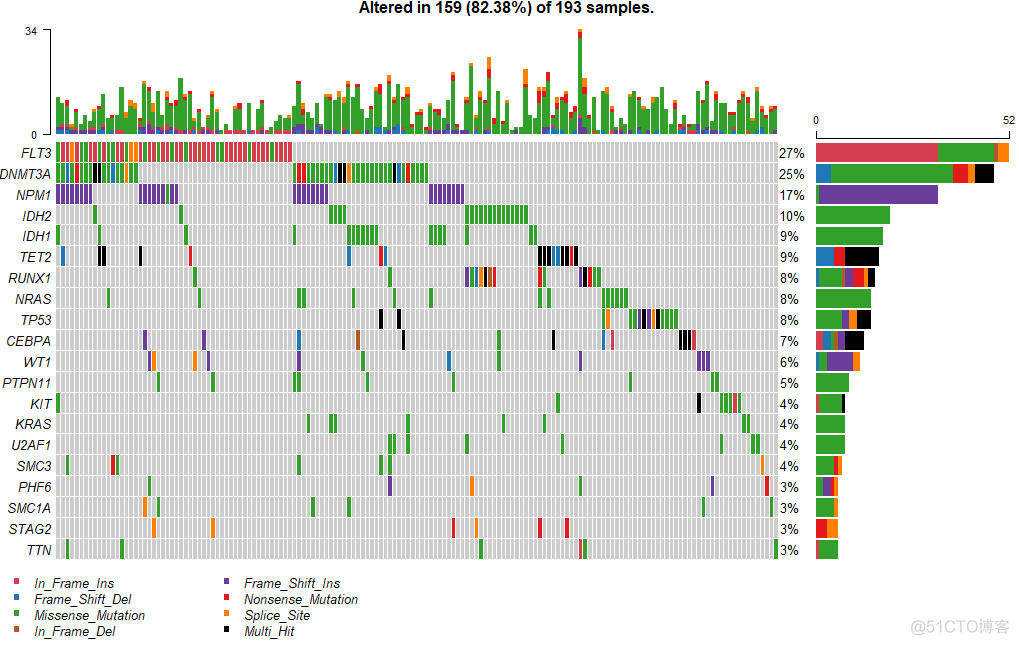
整张图分成了3个部分,第一部分是中间的热图,每一行代表一个基因,每一列代表一个样本,展示了每个样本不同突变类型的分布,第二部分是右侧的堆积柱状图,表示每个基因上不同突变类型位点的频数分布,第三部分是上方的堆积柱状图,表示每个样本中不同突变类型位点的频数分布。
3. oncostrip
和oncoplot类似,只展示其中的热图部分,代码如下
> oncostrip(maf = laml)可视化结果示意如下
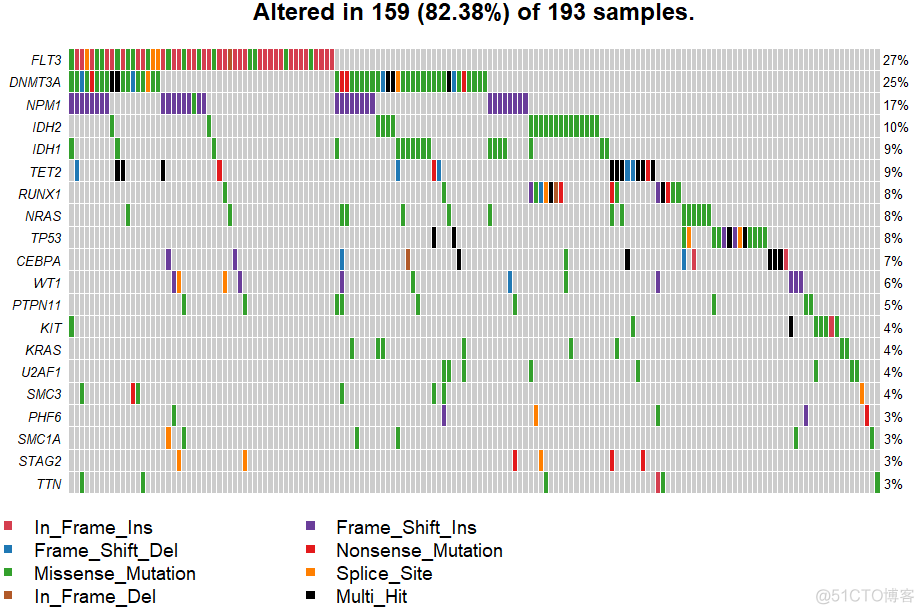
4. 颠换和转换
统计所有SNV中颠换和转换的数量,并进行可视化,代码如下
> laml.titv = titv(maf = laml, plot = FALSE, useSyn = TRUE)> plotTiTv(res = laml.titv)
可视化结果示意如下
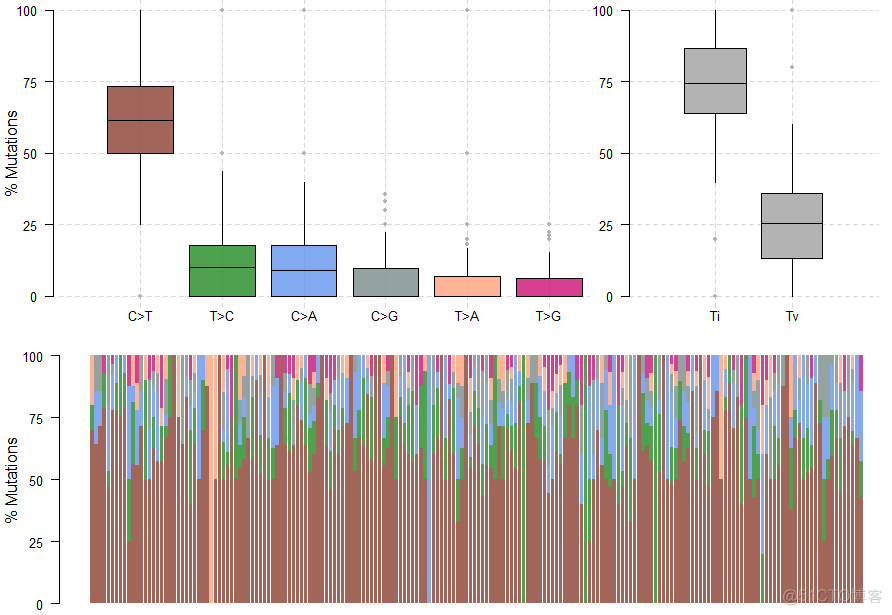
上方的图为每种突变类型以及颠换,转换的位点在所有样本中分布的箱体图,下方的图为每个样本中不同突变类型的百分比堆积柱状图。
5. lollipop plot
lollipop plot用于展示突变对于蛋白质的影响,代码如下
> lollipopPlot(maf = laml, gene = 'DNMT3A', AACol = 'Protein_Change', showMutationRate = TRUE)可视化的结果如下
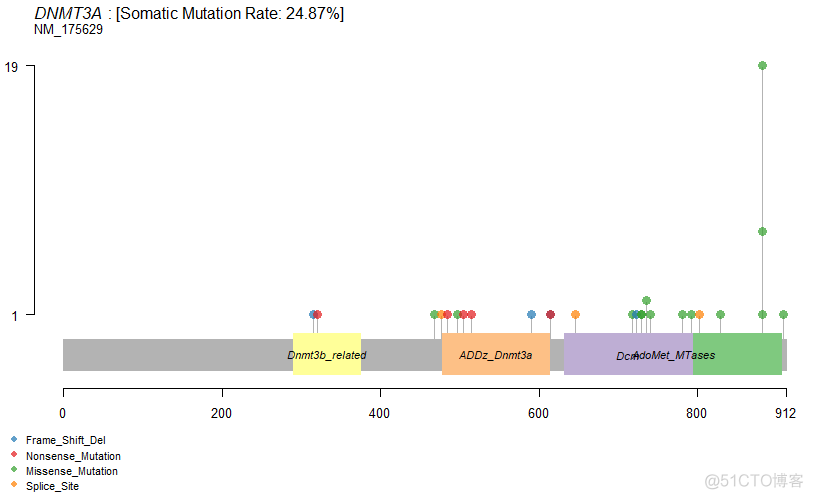
默认情况下,以最长转录本为参考,用圆点表示不同类型变异位点在转录本上的位置,用竖线的高度表示变异位点对应的频率。
6. TCGA compare
用于比较TCGA中不同肿瘤队列间突变频率的分布,代码如下
> laml.mutload = tcgaCompare(maf = laml, cohortName = 'LAML')可视化结果如下
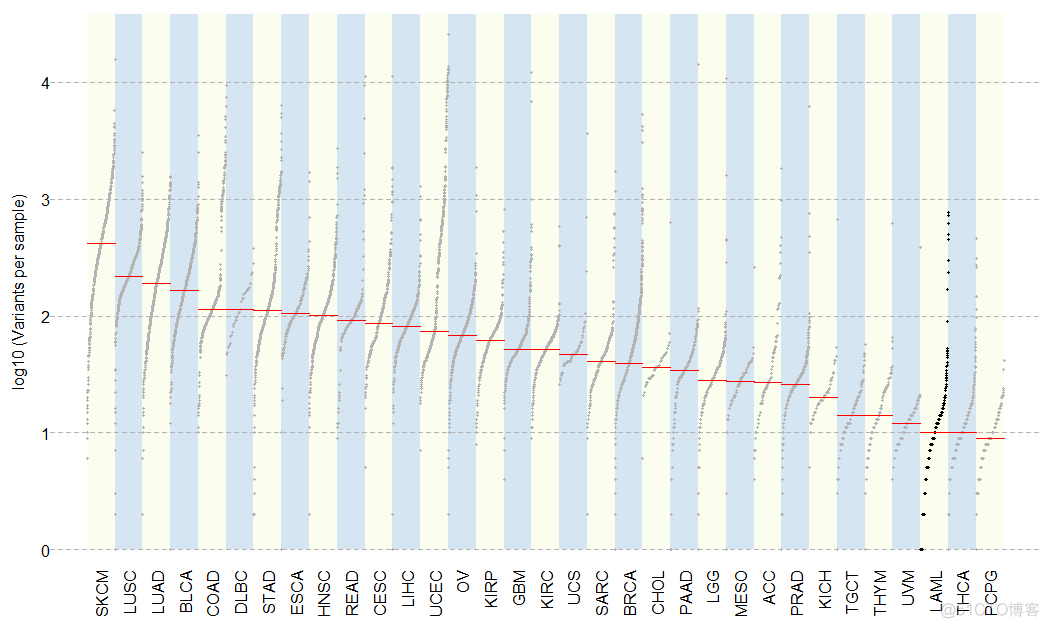
横坐标为肿瘤队列,对应不同的肿瘤,纵坐标为该肿瘤队列中每个样本的体细胞突变个数,红色的线代表中位数。
除了以上功能外,maftools还有更多高级的用法,详情请参考官方文档。
·end·

