目前,较为流行的突变注释软件有以下3种 ANNOVAR SnpEff Variant Effect Predictor(VEP) 这三款软件适用范围广,可以注释任何的基因组变异,无论是germline还是somatic variants。通用性强的同时,
目前,较为流行的突变注释软件有以下3种
这三款软件适用范围广,可以注释任何的基因组变异,无论是germline还是somatic variants。通用性强的同时,带来的问题就是针对肿瘤基因组研究而言,其注释结果中缺乏肿瘤特异性的注释内容,而且其注释结果为VCF格式,需要进一步转换为MAF格式才可以进行肿瘤研究的下游分析,不够便利。
为了解决这个问题,Broad Institute的科学家开发了一款名为oncotator的注释软件,专门针对肿瘤基因组研究进行设计,其注释结果可以直接输出成MAF格式,官网如下
https://software.broadinstitute.org/cancer/cga/oncotator
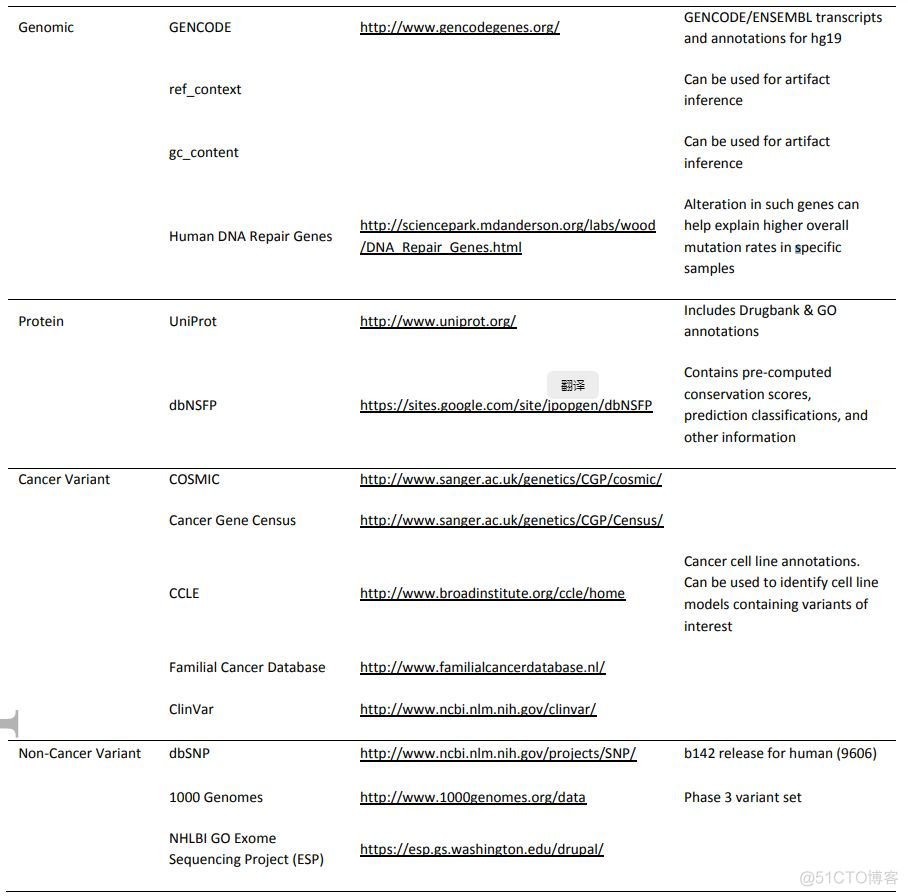
该软件集成了14种不同来源的注释信息,分成以下4大类别
不同类别对应的数据库展示如下
该软件的源代码保存在github上,网址如下
https://github.com/broadinstitute/oncotator/releases
同时该软件依赖一个整合好的数据库,下载方式如下
wget -r "ftp://gsapubftp-anonymous@ftp.broadinstitute.org/bundle/oncotator/"数据库大小为17G, 软件采用python进行开发,其安装过程比较简单,基本用法如下
oncotator \-v \
--db-dir /oncotator_v1_ds_Jan262015 \
--input_format=VCF \
--output_format=TCGAMAF \
input.vcf \
output.maf \
hg19
db-dir参数指定下载的数据数据库的路径,最后三个参数对应输入文件,输出文件和基因组版本,该软件只支持hg19版本。
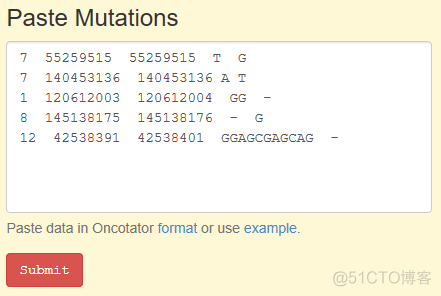
为了方便没有编程经验的科研工作者,同时也提供了web 服务,网址如下
http://portals.broadinstitute.org/oncotator/
上传tsv格式的突变数据即可,示意如下
结果展示如下
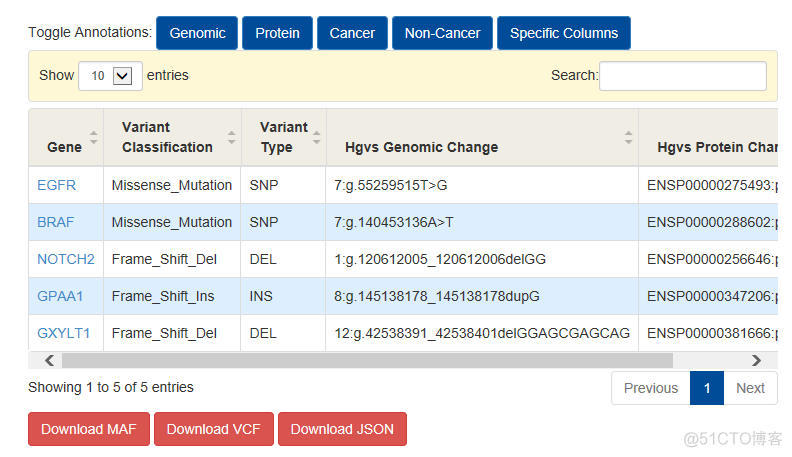
通过该软件可以得到MAF格式的注释文件,更加方便的用于下游的数据分析。
·end·

