network based analysis, 基于网络的数据分析,是目前最常见的研究基因功能的方法,最典型的有pathway和蛋白质相互作用网络的分析,基因间的相互作用构成了一个网络,通过基于网络数据的挖掘算法,挖掘潜在的hub基因。
基因的功能注释来源于数据库,对于pathway而言,有kegg pathway, reactome等数据库,对于ppi而言,有string, IntAct等数据库,不同数据库收录的数据内容和格式都不尽相同,为了更加方便的检索各种数据库,有科学家整合了多个pathway和ppi先关的数据库中的信息,构建了一个综合性的网站,pathway common, 相关的文章发表在Nucleic Acids Research上,链接如下
https://academic.oup.com/nar/article/39/suppl_1/D685/2506078
数据库的网址如下
https://www.pathwaycommons.org/
最初版本整合了包括BioGRID, HPRD, IncAct等共9个pathway和基因interactions的数据库中的信息,详细列表如下
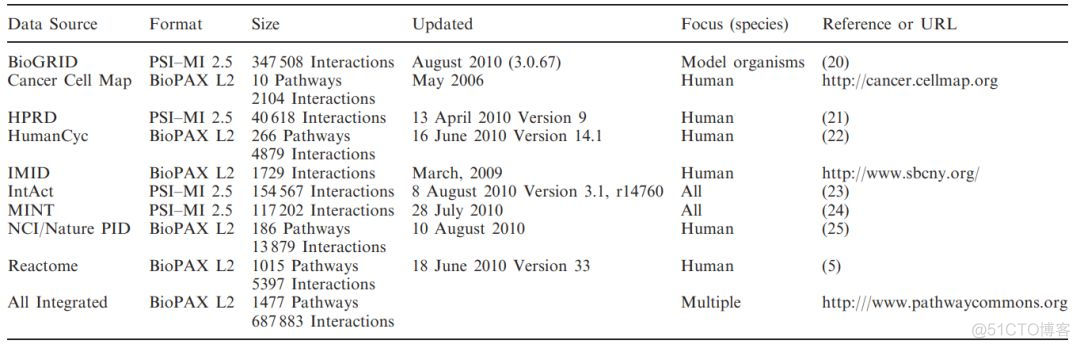
后来又进一步整合了以下数据库中的信息
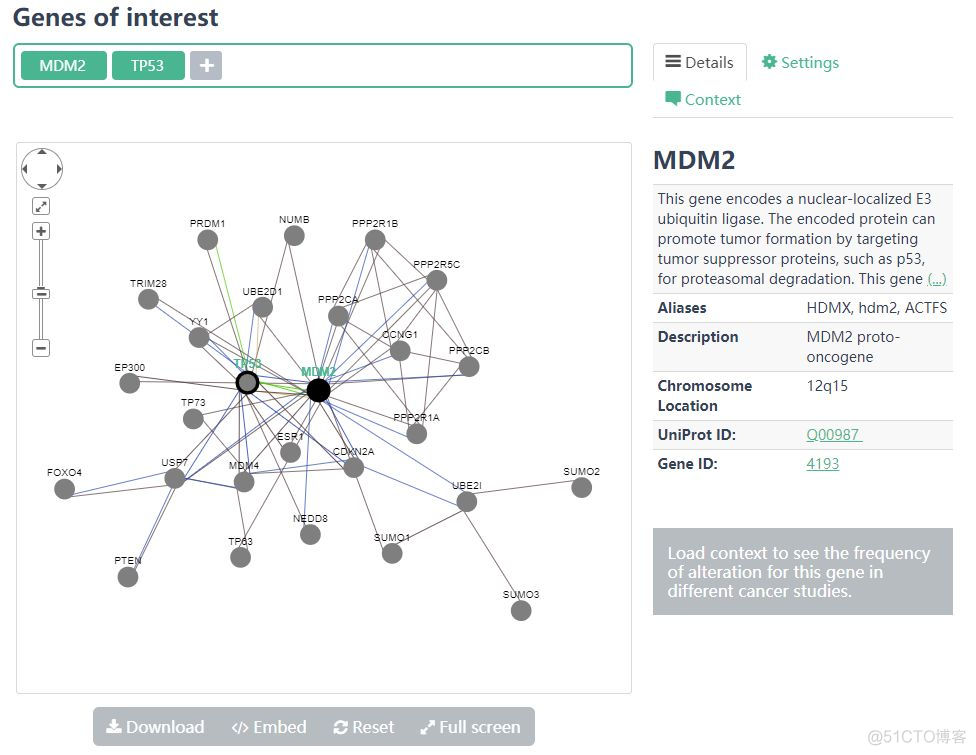
通过首页的Search功能,可以根据pathway或者基因名称来检索数据库中的信息,以TP53为例,结果示意如下
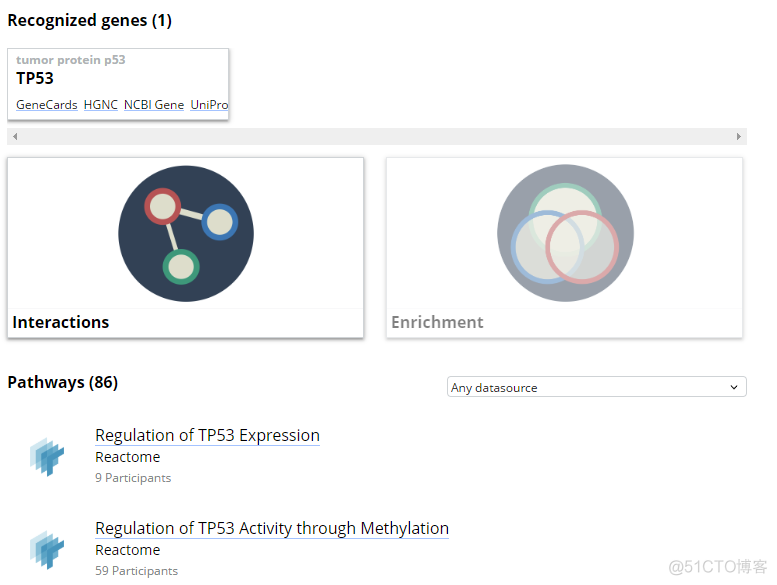
包含了该基因相关的相互作用interactions和pathway两种信息, 示意如下
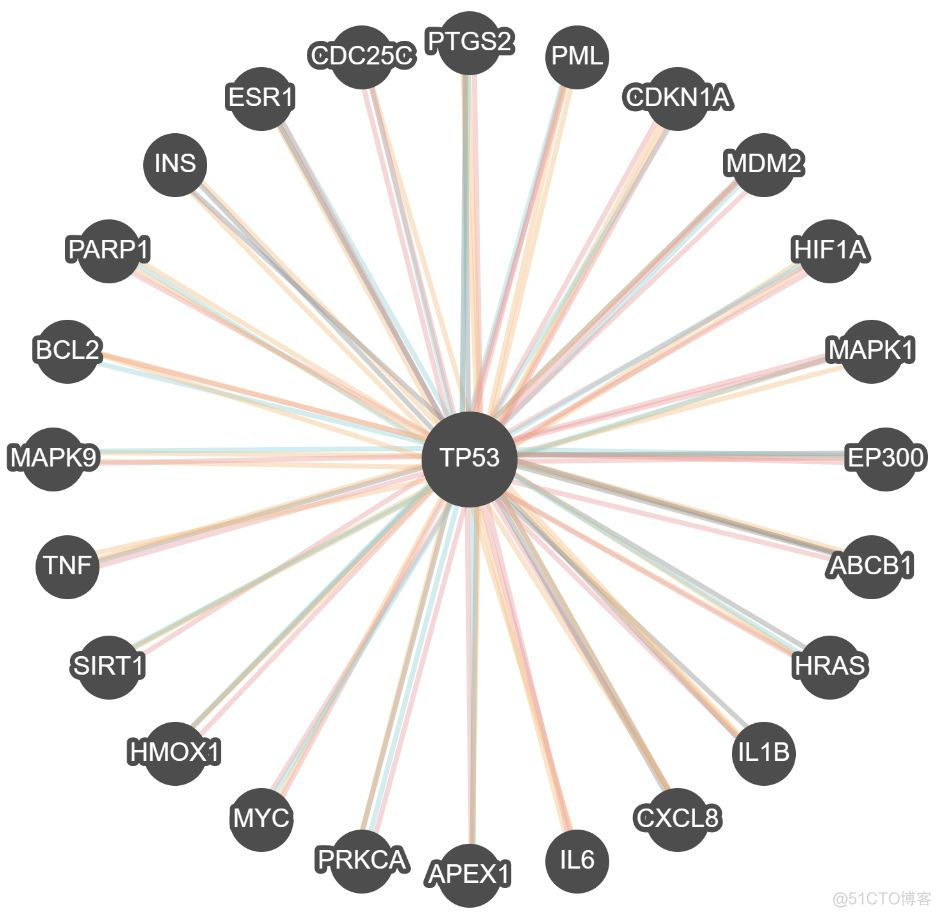
1. interactions
利用各个数据库中收录的基因相互作用信息,绘制如下的基因互作网络
2. Enrichment
如果一次5个以上的基因进行检索,在Enrichment中会展示输入的基因列表所富集的通路,示意如下

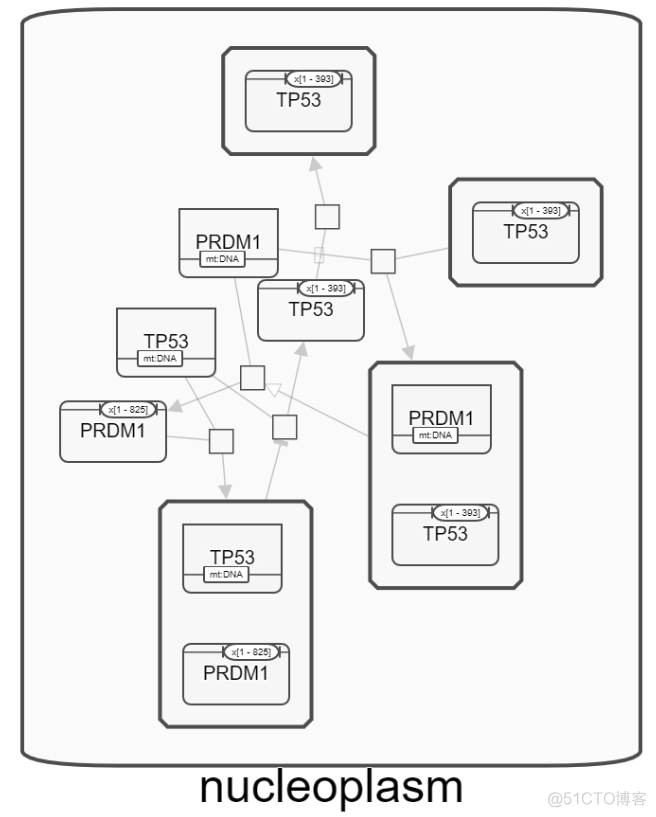
3. pathway
点击每条pathway, 可以查看详细的通路图,示意如下
通过官网的PCViz功能,可以查看多个基因间功能互作网络,结果示意如下
pathway common数据库提供了一个统一的数据交互格式BioPAX, 所有的数据都可以通过以下FTP地址下载得到
https://www.pathwaycommons.org/archives/
通过该数据库,可以方便的开展基于网络的基因功能研究,当然这种整合性数据库在方便的同时也有来源数据库的限制,没有一个数据库可以涵盖所有相关的数据库,比如ppi最常用的string数据库就没有包含在其中,但是对于pathway而言,该数据库算得上是非常全面了。
·end·

