eQTL的分析结果本质就是一些调控基因表达的SNP位点,在结果展示时,最经典的可视化方式如下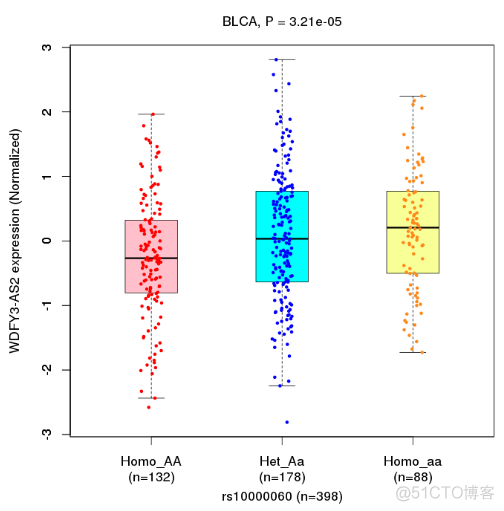
按照SNP分型结果对样本进行分组,用箱体图或者小提琴图的方式展示不同组别中基因表达量的分布,直观的比较不同分组中表达量的差异。除此之外,也可以借鉴GWAS分析结果的展示形式,绘制曼哈顿图和QQ图,在以下文章中,就采用了曼哈顿图来展示eQTL的结果
Genomics of ADME gene expression: Mapping expression quantitative trait loci relevant for absorption, distribution, metabolism and excretion of drugs in human liver
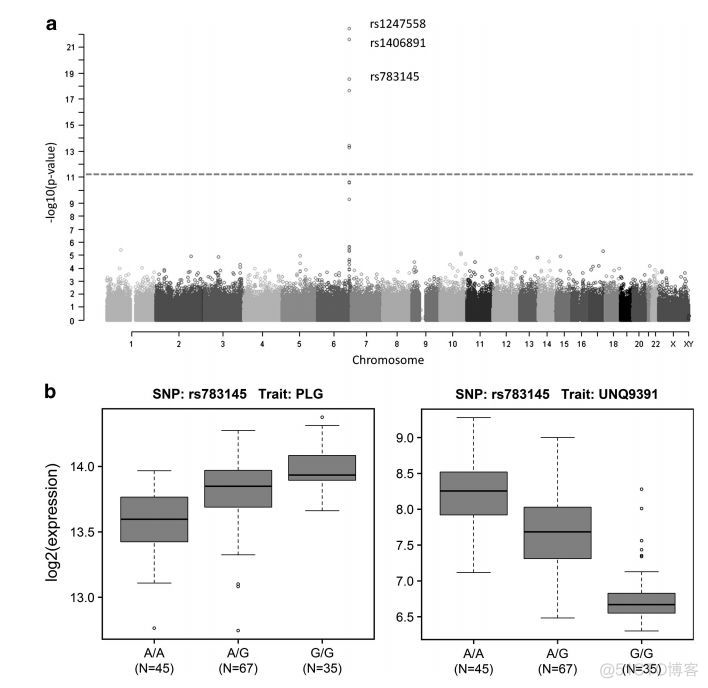
以上方法都是针对单个study中的eQTL分析结果,当我们想要比较多个study的结果时,比如比较不同组织中eQTL分布的差异,就需要新的展现形式了。此时,有一个网站值得借鉴,那就是WormQTL, 网址如下
http://www.bioinformatics.nl/EleQTL
该网站用来展示秀丽隐杆线虫的eQTL分析结果,是在AraQTL(一个展示拟南芥eQTL结果的网站)的基础上发展而来的,采用了eQTL位点的LOD值来表征eQTL profile, 从而可以方便的展示和比较多个study的结果,有以下几种可视化形式
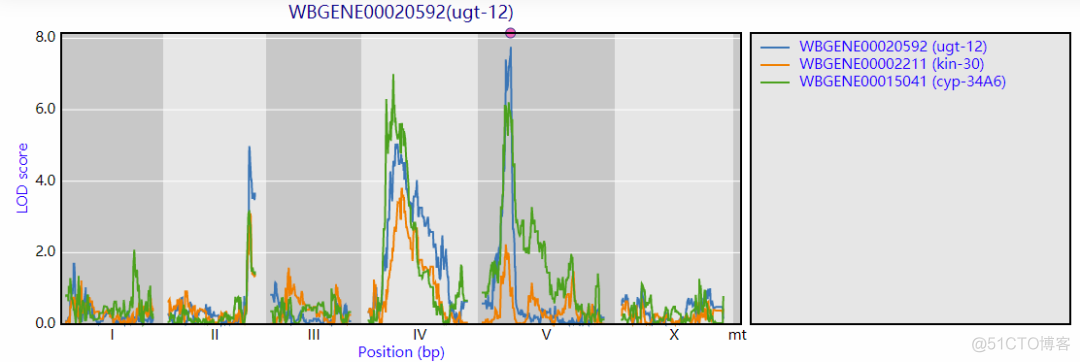
1. Gene eQTL profiles
对于一个gene而言,在全基因组范围内,其eQTL位点是很多的,将每个位点的染色体位置作为横坐标,LOD值作为纵坐标,可以绘制如下所示的折线图
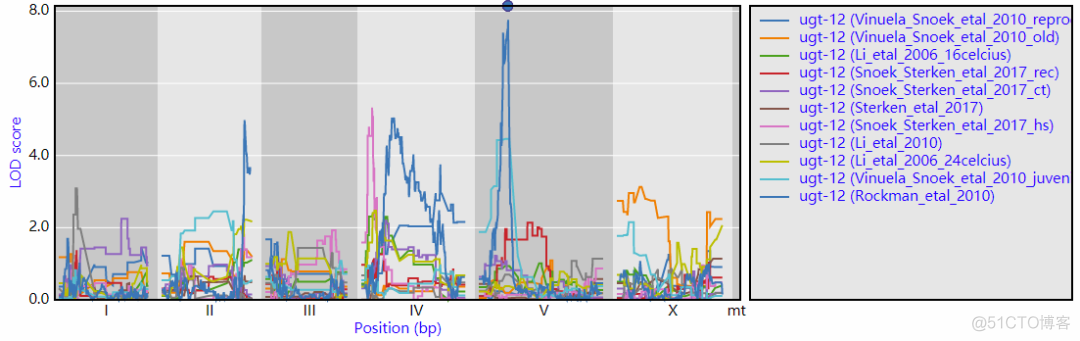
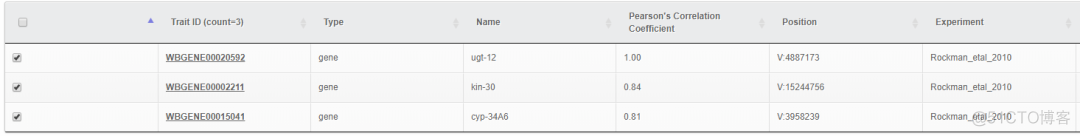
每条折线代表一个study的eQTL结果,不同study用不同颜色表示。对应的表格数据如下
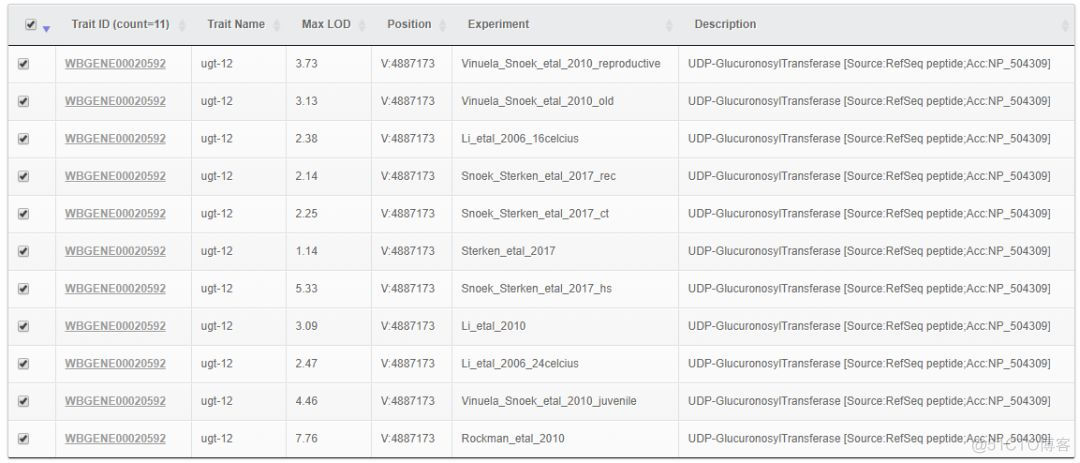
在eQTL分析中,Trait对应的就是一个gene,LOD值最大的位点认为是统计学最显著的eQTL位点。
2. Correlated eQTL profiles
利用eQTL的LOD值,可以计算不同study或者不同基因的eQTL之间的相关性,该网站采用了泊松相关系数来计算不同eQTL profiles之间的相关性,结果如下
3. Cis-trans plot
采用散点图的形式展示所有eQTL-gene对在染色体上的分布情况,示意如下

图中每个点代表一个eQTL-gene对,点的颜色根据LOD值的大小进行映射,从负到正,颜色从红色过渡到蓝色。
该网站还有更多的可视化用法,具体其参考其官方文档。
·end·

生物信息入门
只差这一个
公众号
