驱动基因的识别是肿瘤基因组学研究中的一项重要内容,NCG是一个肿瘤驱动基因的数据库,目前最新版本为v6.0, 网址如下 http://ncg.kcl.ac.uk/index.php 共收录了2372个驱动基因,分
驱动基因的识别是肿瘤基因组学研究中的一项重要内容,NCG是一个肿瘤驱动基因的数据库,目前最新版本为v6.0, 网址如下
http://ncg.kcl.ac.uk/index.php
共收录了2372个驱动基因,分成以下两个部分
对于每个基因,提供了多种注释信息,以PTEN为例,包含以下几个部分的注释信息
1. General information
提供了基因在NCBI, Ensembl, Uniprot等数据库的链接,同时也提供了蛋白结构域SMART, 体细胞突变COSMC,相关疾病OMIM等数据库的链接,示意如下
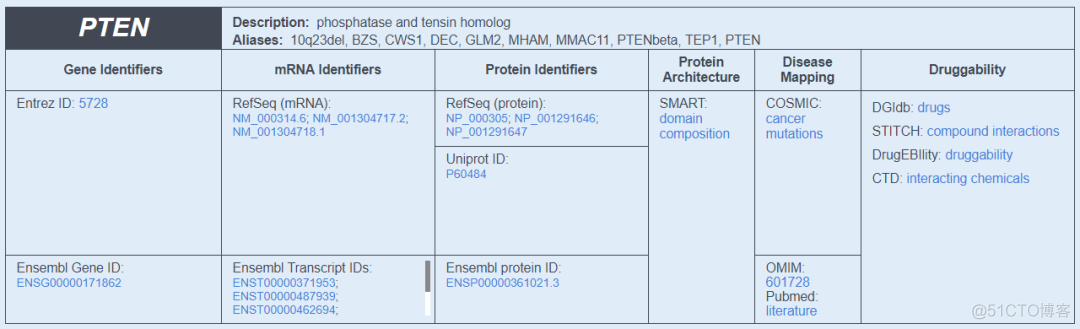
2. Cancer Information
提供了该基因相关的肿瘤信息,以COSMIC数据库和对应的pubmed文献链接的形式查看,示意如下
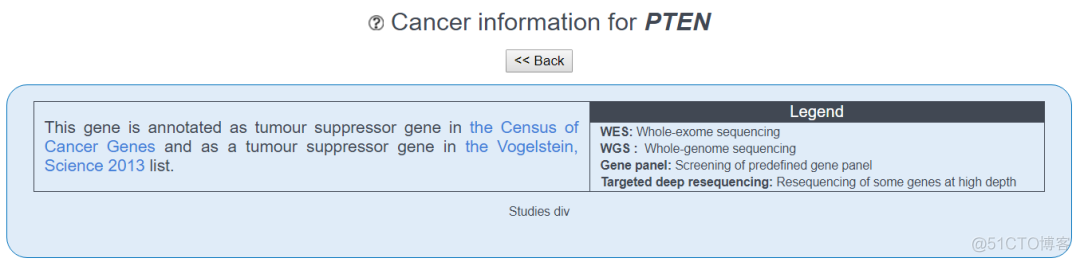
3. Orthology
提供了该基因在不同物种间的同源信息,示意如下
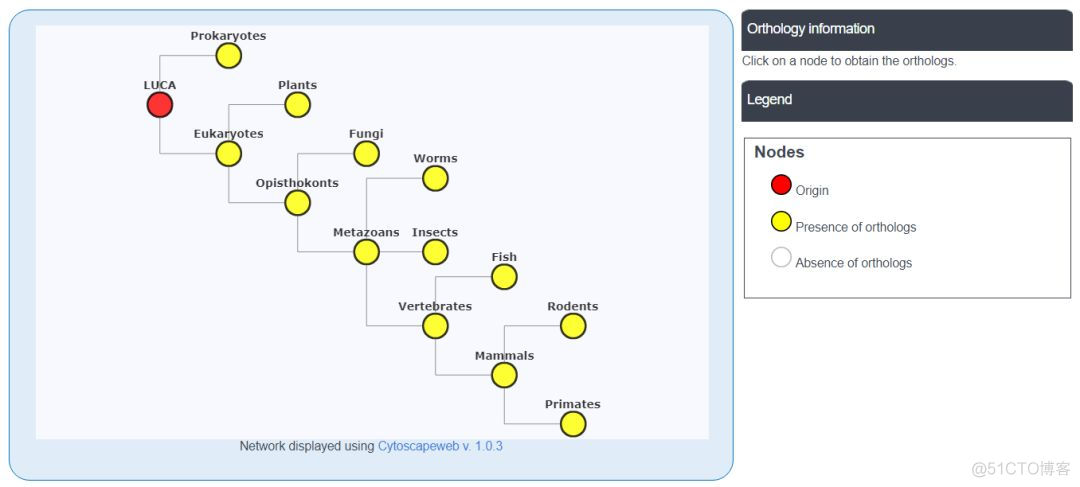
4. Gene Expression in Normal Tissues
提供了来自以下两个数据库中的基因表达量信息
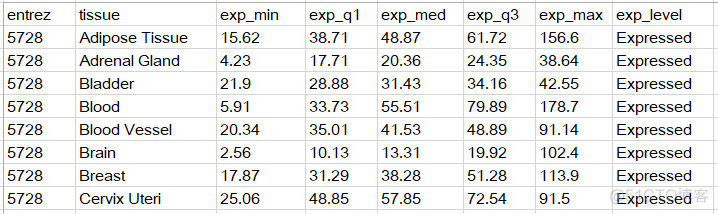
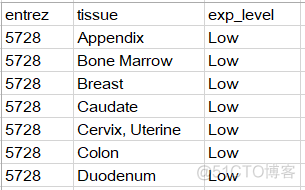
5. Protein Expression in Normal Tissues
提供了来自HPA数据库中的蛋白质表达量信息,结果示意如下
6. Protein Function
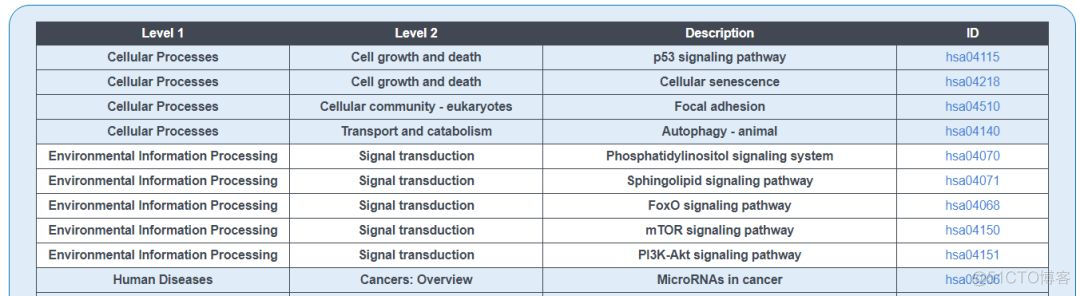
提供了来自以下3个数据库的功能注释
结果示意如下
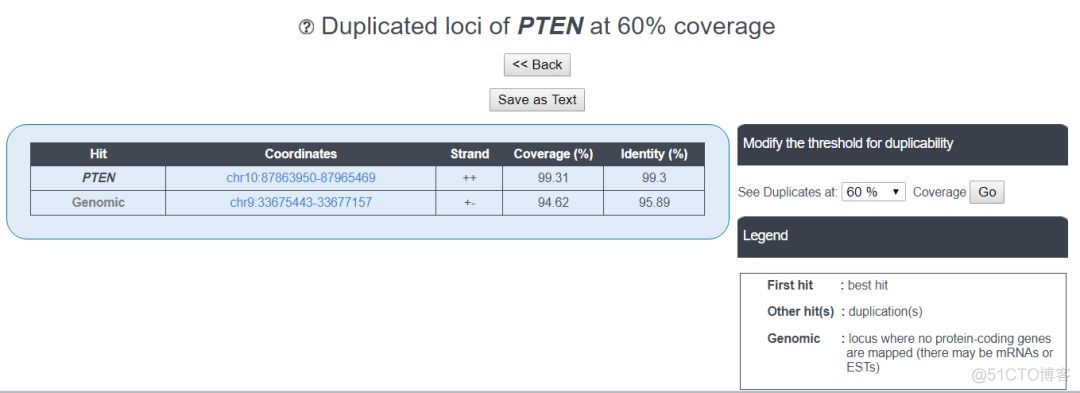
7. Duplicablity
通过blat比对的方式鉴定在该基因在基因组上的同源区域,结果示意如下
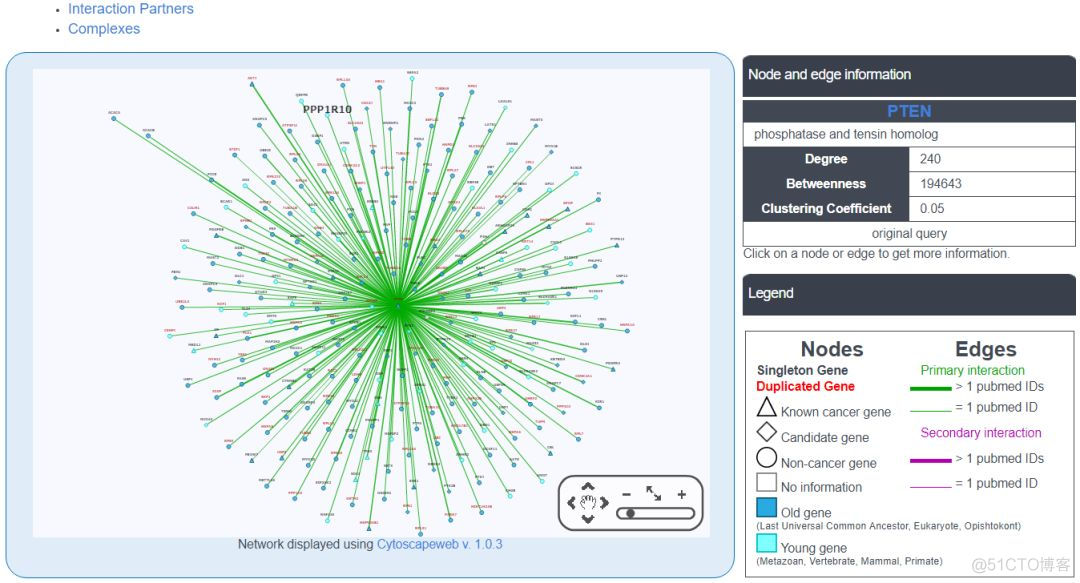
8. Network Properties
利用BioGRID, MIntAct, DIP, HPRD, Reactome等数据库,提供了该基因与其他基因的互作网络,由蛋白互作和蛋白复合体两部分构成,结果示意如下
9. Gene Expression in Cancer Cell Lines
提供了来自CCLE数据库的基因表达量信息,结果示意如下
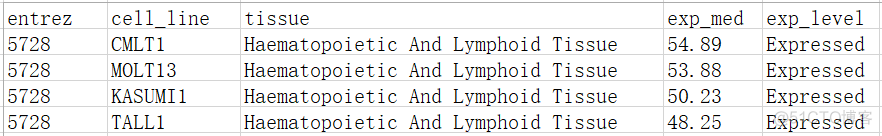
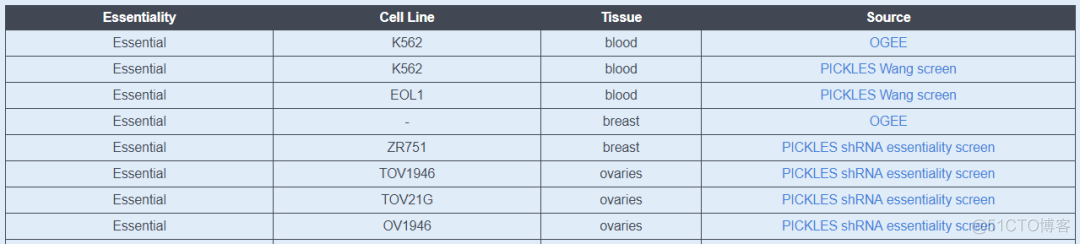
10. Gene Essentiality
利用PICKLES, OGEE数据库对基因的重要程度进行评分,结果示意如下
11. miRNA-Gene interactions
利用miRTarBase, miRecords 数据库中的miRNA靶基因信息,提供了该基因与miRNA的调控网络,结果示意如下
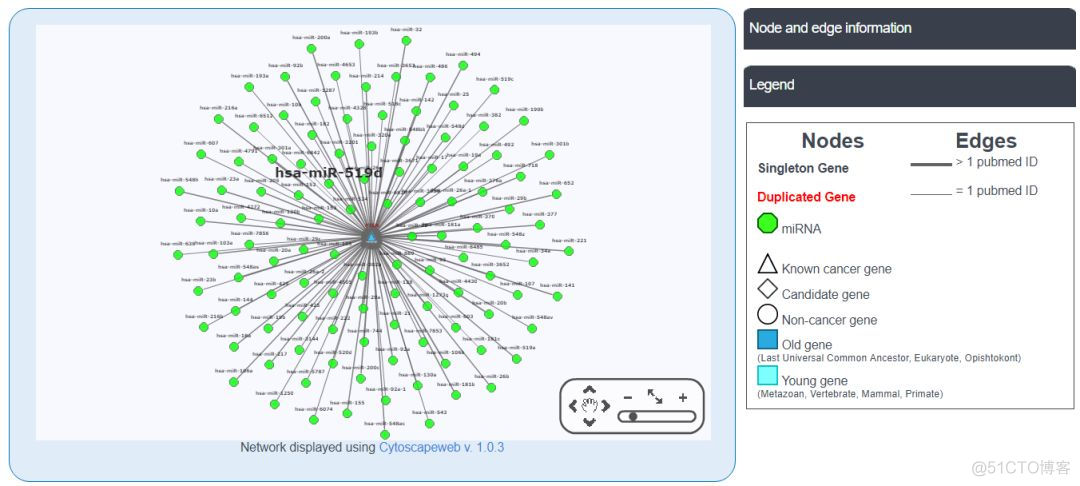
通过该数据库,可以快速检索肿瘤的驱动基因,并对基因的各种注释信息进行查看和分析。
·end·

