在之前meta分析的文章中我们介绍了森林图的画法,典型的森林图如下所示
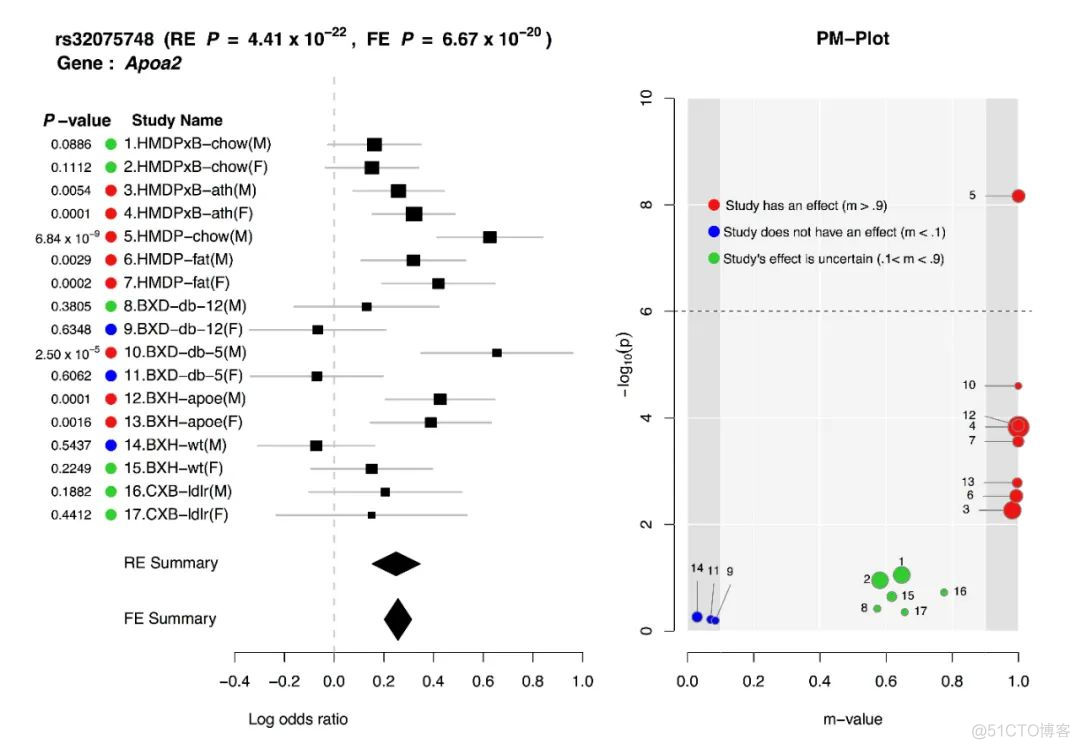
每一行表示一个study,用errorbar展示log odds ratio值的分布,并将p值和m值标记在图中。森林图主要用于多个study的分析结果的汇总展示。
在构建预后模型时,通常会先对所有基因进行单变量cox回归,然后筛选其中显著的基因进行多变量cox回归来建模,对于cox回归的结果,每个基因也都会有一hazard ratio和对应的p值,也可以用森林图的形式来展现,比如NAD+的文献中就采用了这样的一张森林图
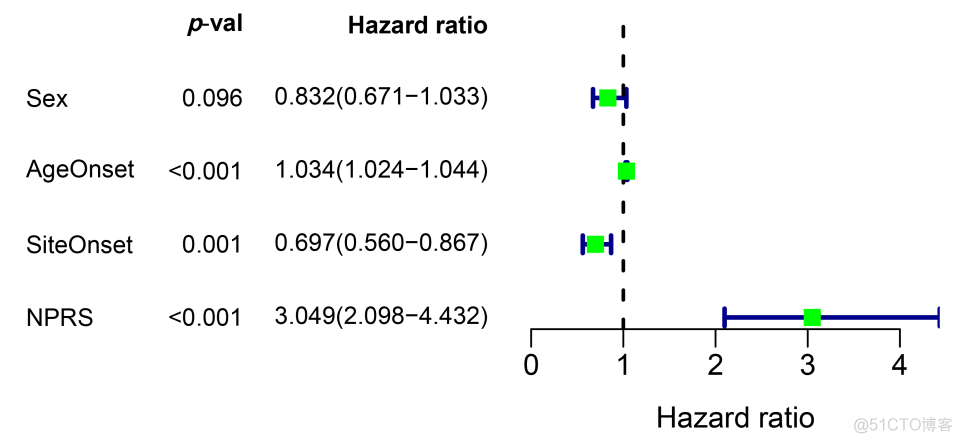
每一行表示一个变量,用errorbar展示该变量对应的风险值的大小和置信区间,并将风险值和p值标记在图上。
根据cox生存分析的结果绘制森林图有多种方式,使用survminer包的ggforest函数,是最简便的一种,代码如下
> require("survival")
> model <- coxph( Surv(time, status) ~ sex + rx + adhere,
+ data = colon )
> model
Call:
coxph(formula = Surv(time, status) ~ sex + rx + adhere, data = colon)
coef exp(coef) se(coef) z p
sex -0.04615 0.95490 0.06609 -0.698 0.484994
rxLev -0.02724 0.97313 0.07690 -0.354 0.723211
rxLev+5FU -0.43723 0.64582 0.08395 -5.208 1.91e-07
adhere 0.29355 1.34118 0.08696 3.376 0.000736
Likelihood ratio test=46.51 on 4 df, p=1.925e-09
n= 1858, number of events= 920
> ggforest(model)
效果图如下
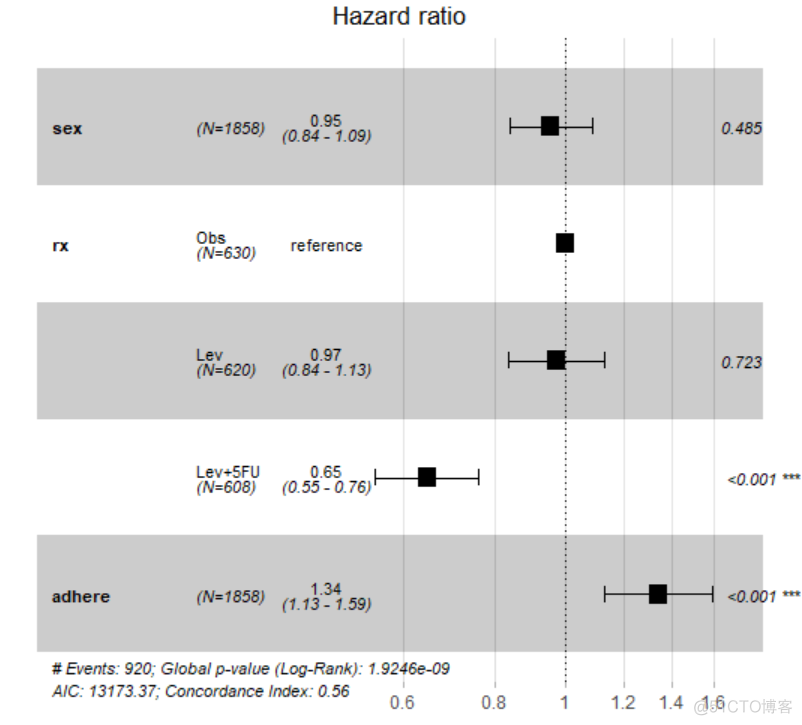
这种方式确实出图简单,一步到位,但是提供的参数却很少,灵活性很小,基本上没法修改图中的元素,另外一种方式,就是使用forest这个R包,这个R包灵活性很大,通过调参可以实现很多自定义效果,基本用法如下
> test_data <- data.frame(
+ coef = c(1.59, 1.24),
+ low = c(1.4, 0.78),
+ high = c(1.8, 1.55)
+ )
> forestplot(
+ labeltext = row_names,
+ mean = test_data$coef,
+ lower = test_data$low,
+ upper = test_data$high,
+ zero = 1,
+ cex = 2,
+ lineheight = "auto",
+ xlab = "Lab axis txt"
+ )
效果图如下
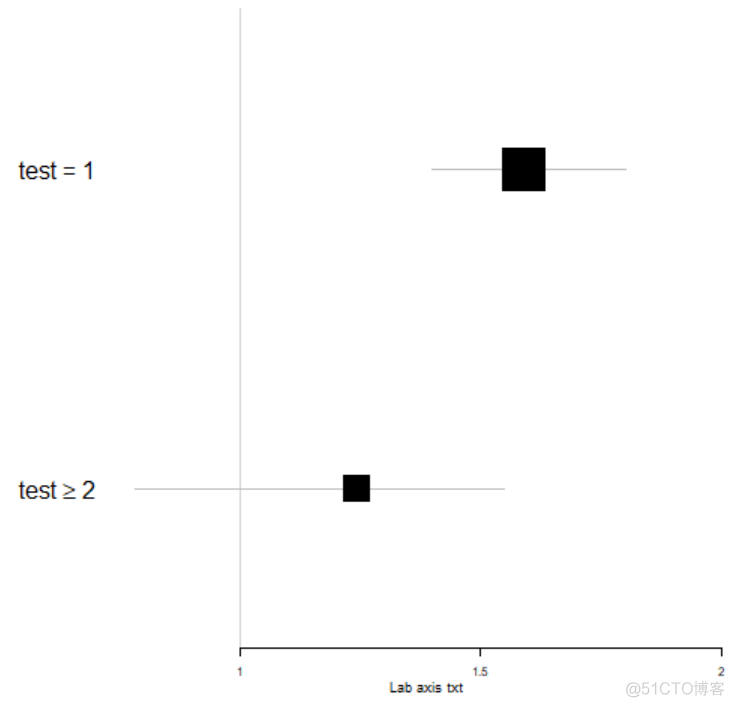
虽然输出很简陋大,但是从基本用法可以看出,我们可以自定义变量名称,指定风险值的大小,这样我们只需要从cox回归的结果中提取我们需要绘图的元素进行绘制即可。
基本用法之外中添加的变量是单列注释,如果要实现文献中图片的多列注释效果,可以参考下面这个例子
> test_data <- data.frame(+ coef1 = c(1, 1.59, 1.3, 1.24),
+ coef2 = c(1, 1.7, 1.4, 1.04),
+ low1 = c(1, 1.3, 1.1, 0.99),
+ low2 = c(1, 1.6, 1.2, 0.7),
+ high1 = c(1, 1.94, 1.6, 1.55),
+ high2 = c(1, 1.8, 1.55, 1.33)
+ )
>
> col_no <- grep("coef", colnames(test_data))
> row_names <- list(
+ list("Category 1", "Category 2", "Category 3", expression(Category >= 4)),
+ list(
+ "ref",
+ substitute(
+ expression(bar(x) == val),
+ list(val = round(rowMeans(test_data[2, col_no]), 2))
+ ),
+ substitute(
+ expression(bar(x) == val),
+ list(val = round(rowMeans(test_data[3, col_no]), 2))
+ ),
+ substitute(
+ expression(bar(x) == val),
+ list(val = round(rowMeans(test_data[4, col_no]), 2))
+ )
+ )
+ )
>
> coef <- with(test_data, cbind(coef1, coef2))
> low <- with(test_data, cbind(low1, low2))
> high <- with(test_data, cbind(high1, high2))
> forestplot(row_names, coef, low, high,
+ title = "Cool study",
+ zero = c(0.98, 1.02),
+ grid = structure(c(2^-.5, 2^.5),
+ gp = gpar(col = "steelblue", lty = 2)
+ ),
+ boxsize = 0.25,
+ col = fpColors(
+ box = c("royalblue", "gold"),
+ line = c("darkblue", "orange"),
+ summary = c("darkblue", "red")
+ ),
+ xlab = "The estimates",
+ new_page = TRUE,
+ legend = c("Treatment", "Placebo"),
+ legend_args = fpLegend(
+ pos = list("topright"),
+ title = "Group",
+ r = unit(.1, "snpc"),
+ gp = gpar(col = "#CCCCCC", lwd = 1.5)
+ )
+ )
效果图如下
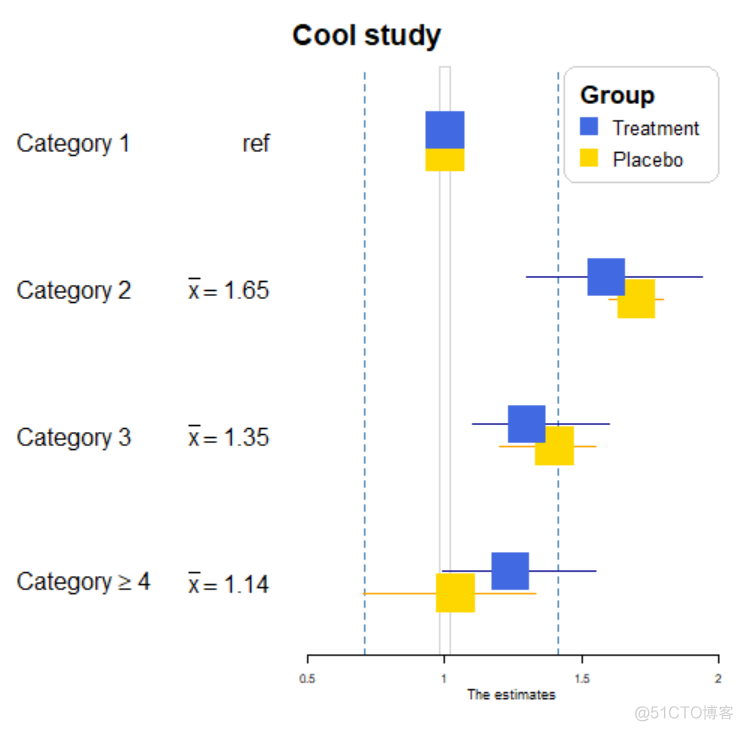
基于上述知识储备和函数的帮助文档,我们就可以实现和文章中图片一致的效果图了,只需要仔细钻研函数的帮助文档即可。
·end·
—如果喜欢,快分享给你的朋友们吧—
原创不易,欢迎收藏,点赞,转发!生信知识浩瀚如海,在生信学习的道路上,让我们一起并肩作战!
本公众号深耕耘生信领域多年,具有丰富的数据分析经验,致力于提供真正有价值的数据分析服务,擅长个性化分析,欢迎有需要的老师和同学前来咨询。
写在最后

