生存分析在预后建模中的作用不必多言,在之前介绍的NAD+基因的文献中,出现了3种生存分析的可视化方式,文献链接如下 https://www.frontiersin.org/articles/10.3389/fcell.2022.831273/full
生存分析在预后建模中的作用不必多言,在之前介绍的NAD+基因的文献中,出现了3种生存分析的可视化方式,文献链接如下
https://www.frontiersin.org/articles/10.3389/fcell.2022.831273/full
对于生存分析的可视化,最常用的就是R包survminer, 核心函数就是ggsurvplot,链接如下
https://rpkgs.datanovia.com/survminer/index.html
下面介绍下如何通过这个R包来实现可视化的具体代码
1. 第一种模式
文献中图示如下
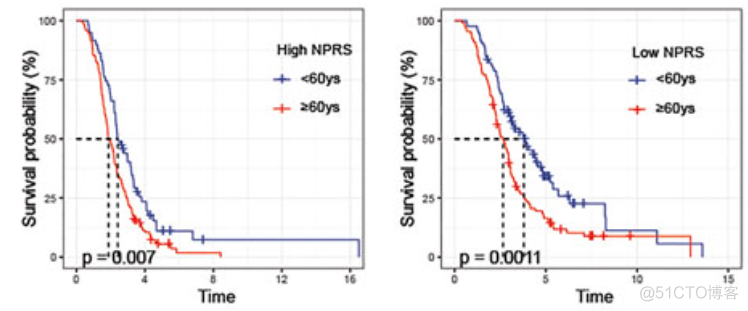
在传统的生存曲线的基础上,标记了p值。首先我们用默认参数先得到一张最基本的生存曲线图
> library("survminer")> require("survival")
> fit <- survfit(Surv(time, status) ~ sex, data = lung)
> head(lung)
inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
1 3 306 2 74 1 1 90 100 1175 NA
2 3 455 2 68 1 0 90 90 1225 15
3 3 1010 1 56 1 0 90 90 NA 15
4 5 210 2 57 1 1 90 60 1150 11
5 1 883 2 60 1 0 100 90 NA 0
6 12 1022 1 74 1 1 50 80 513 0
> fit
Call: survfit(formula = Surv(time, status) ~ sex, data = lung)
n events median 0.95LCL 0.95UCL
sex=1 138 112 270 212 310
sex=2 90 53 426 348 550
> ggsurvplot(fit, data = lung)
效果图如下
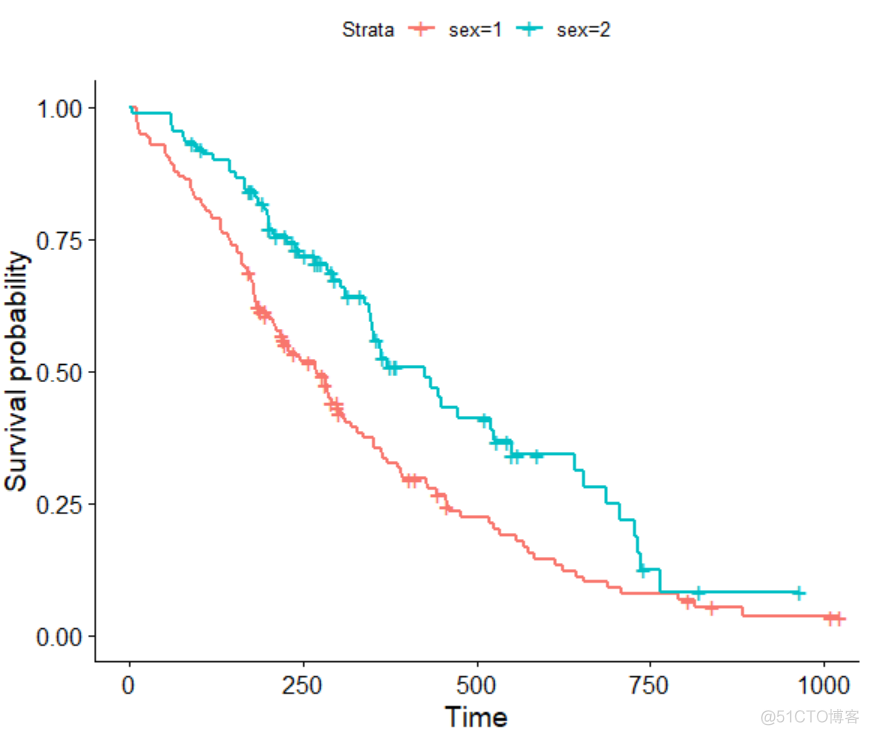
在此基础上,需要做如下调整
1. 调整y轴标题
2. 调整图例的文字
3. 调整不同分组的颜色
4. 添加中位数的直线和p值
5. 调整画框的主题
代码如下
