和GEO数据库类似,ArrayExpress是属于EBI旗下的公共数据库,用于存放芯片和高通量测序的相关数据,网址如下
>https://www.ebi.ac.uk/arrayexpress/
数据来源于下图所示的两个部分
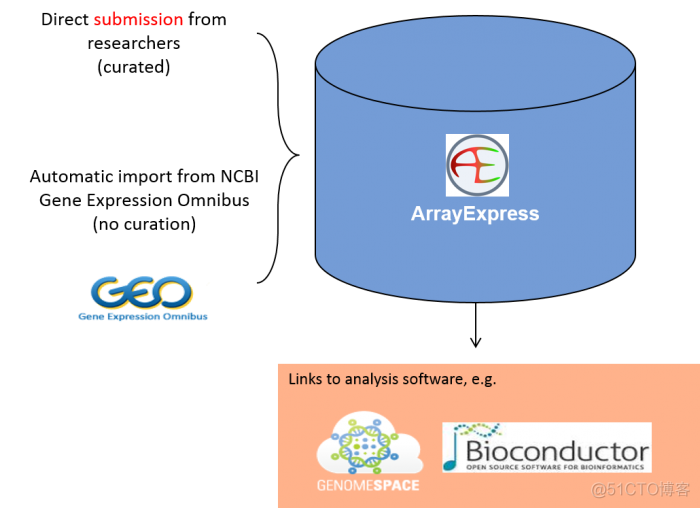
第一部分是由科研工作者提交的数据,第二部分是从GEO数据库自动导入的数据。在NAD代谢相关的文章中,就提到了来自该数据库的原始数据,链接如下
https://www.ebi.ac.uk/arrayexpress/experiments/E-TABM-940/
对于每个数据集,都提供了该数据的简要描述,比如物种,样本数目,平台等信息
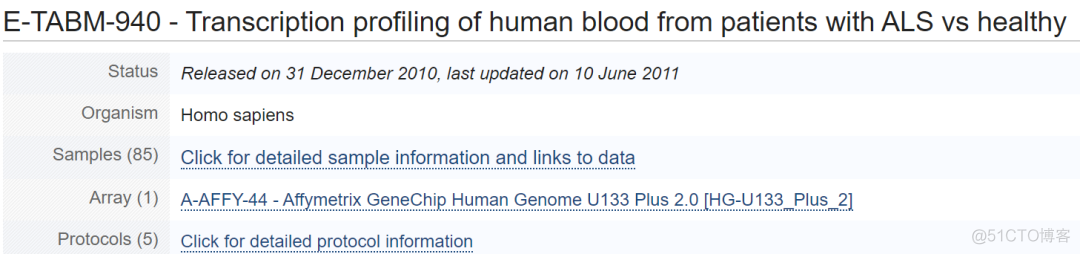
在Files一栏中,可以查看需要下载的文件

点击all available files, 就会显示所有的文件,主要分成了两部分,数据集的原始数据和芯片平台的注释信息
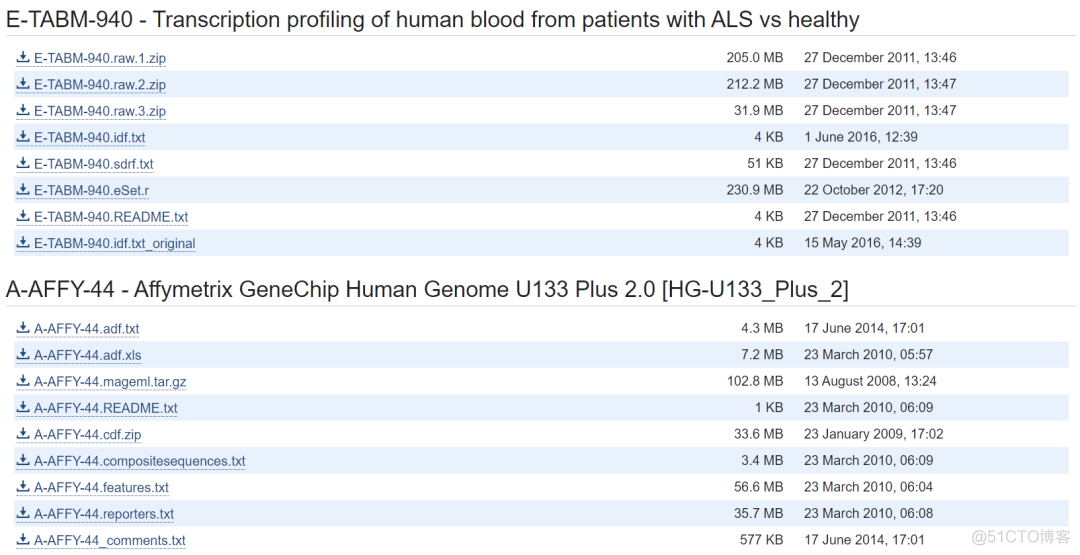
从该数据集的描述可以看到,使用的是Affymetirx的芯片,所以在下载数据集,我们需要获取以下3种信息
1. 芯片表达谱
表达谱包括了原始数据和整理好的表达量文件,在该数据集中,raw.zip就是原始的芯片下机数据。解压缩之后可以看到后缀为cel的原始数据
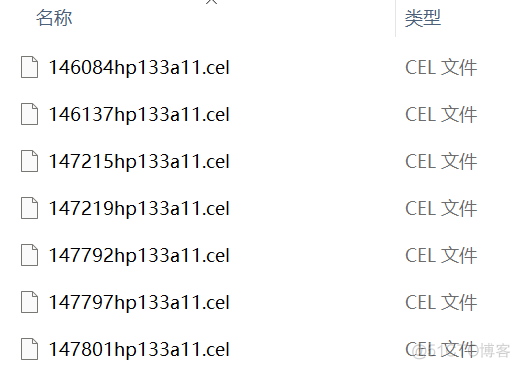
可以通过affy等R包读取这些原始文件,从而生成表达量矩阵。为了方便,该数据集之类提供了一个r对象,即后缀为eSet.r的文件,该文件其实就是读取原始数据后生成的R对象。在R环境中,通过如下代码可以读取该文件
load("E-TABM-940.eSet.r")# 所有的对象默认都叫做study, 所以通过重命名来区分不同的数据集
data <- study
# 查看表达量
head(exprs(data[1:5, 1:5]))
在后缀为idf.txt的文件中,提供了该数据集的一些简要描述信息

在后缀为sdrf.txt的文件中,提供了样本的描述信息,该文件为纯文本文件,每一行为一个样本,部分列名如下
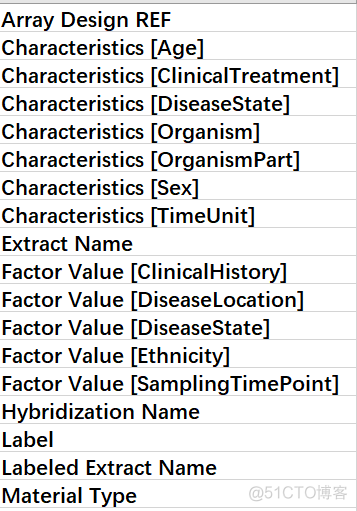
可以看到,包含了样本的各种信息,以Characteristics 和 Factor 开头的列需要重点关注,这里面可能就包含了重要的分组信息和生存数据。
2. 芯片的注释信息
在芯片的数据中,后缀为adf.txt的文件提供了探针的各种注释信息,每一行为一个探针,部分内容示例如下
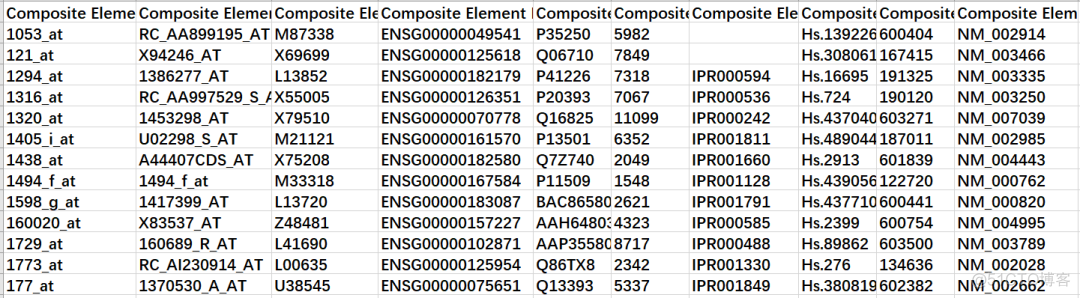
详细的表头如下
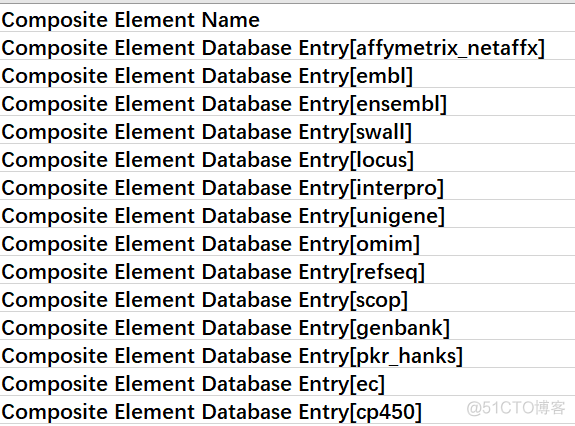
可以看到,包含了ensembl, refseq, genebank等多种注释信息。
另外还有一个非常重要的文件,就是后缀为cdf.gz的文件。在用R读取该文件芯片的原始数据cel文件时,需要对应的cdf文件。对于一些常规型号的芯片,在R中集成了对应的cdf文件,在读取阶段会自动下载对应的cdf文件,但是对于非常规,少见的自定义芯片,就必须手动下载该文件了。
值得注意的是,本文介绍的方法是针对affymetrix芯片平台的,目前芯片平台主要由Agilent, illumina, Affymetirx 3种,不同的芯片来自不同的厂家,其文件格式和处理方式有很大不同,必须针对不同的平台采取不同的方法。
·end·

一个只分享干货的
生信公众号
