在NAD+代谢相关基因的文章中,针对来自GEO数据库的ALS患者的表达谱数据进行了挖掘,本文就以这两批GEO数据为例,来详细展示原始数据的下载过程
公共数据库的数据挖掘有多种模式,每种模式需要的原始数据也不尽相同。所以,首先我们要明确目标,对于这个基因建模而言,我们需要以下两种数据
1. 基因表达谱
2. 患者的生存数据
明确目标之后,我们就可以针对的来分析寻找数据了
1. 基因表达谱
在GEO数据库中收录的表达谱数据有两种类型
1. 基于NGS的表达谱
2. 基于芯片的表达谱
二者的模式稍有不同,芯片平台以探针为单位,而NGS的数据直接以基因为单位。我们最终希望得到的表达谱数据就是行为基因,列为样本的表达谱,所以
1.对于芯片平台,在下载表达谱的同时,我们还需要下载芯片对应平台的注释文件,主要是提供探针到基因的映射关系
2.对于NGS, 基因定量的坐标来自于GTF文件,芯片平台在NGS就对应了GTF的版本
GEO一开始就是用来存储芯片表达谱数据的,所以对于芯片恶言,会有一个通用的格式,以GSE112676为例,数据链接如下
https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE112676
我们需要重点关注两部分
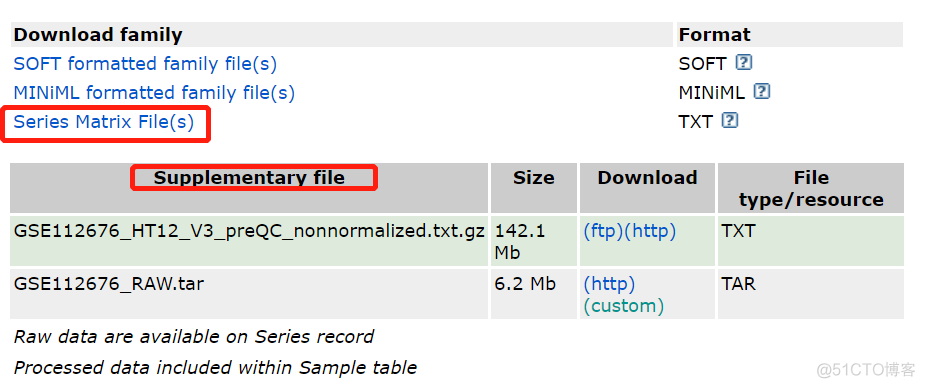
首先看supplementary部分,这部分会提供作者整理好的一些数据,我们可以根据文件名判断是否是我们需要的文件,比如这里的preQC_nonnormalized.txt 就是归一化之后的表达谱,我们直接下载这个文件就可以了。raw.rar 文件是芯片的原始文件,当实在找不到现成的表达谱时,也可以从原始数据重头开始分析。
第二部分关注Download family这部分,该部分提供的是芯片的原始表达谱和样本注释信息,对应3种不同格式,其中的series matrix 是TXT格式,我们使用起来最方便,直接点击链接进去下载即可。
下载完成之后是一个压缩文件,直接解压即可,用excel打开该文件,可以在找到如下图所示的部分数据
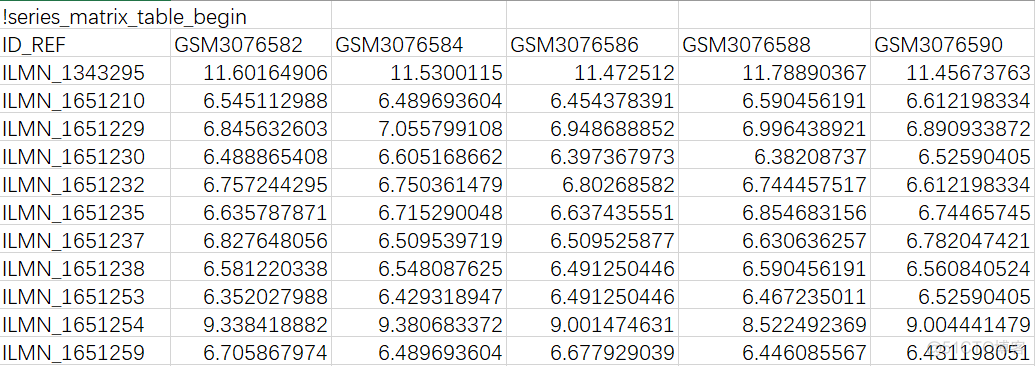
该数据就是探针水平的原始表达量数据了。对于芯片而言,所用的芯片平台都会给出,在platform一栏中可以看到

点击对应链接,可以跳转到该平台的注释信息,示例如下
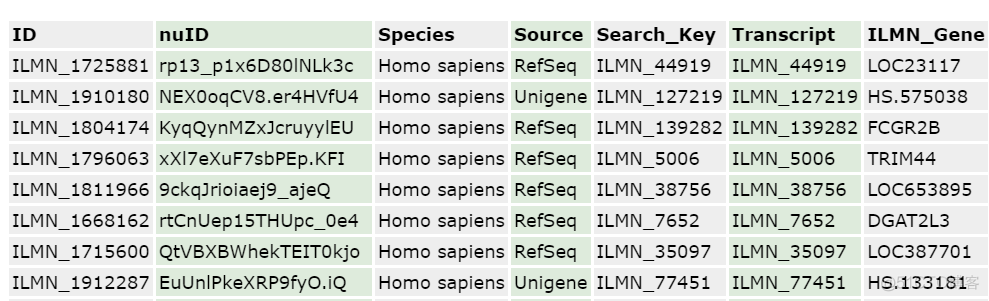
提供了探针对转录本,基因等多种注释信息的映射,我们可以下载完整的注释表格。
2. 生存数据
series matrix文件不仅包含探针表达量信息,在Sample_characteristics_chr1 部分的注释信息中,还会提供关于样本的各种信息,有些作者会把样本的生存数据存放在这里,GSE112676示例如下
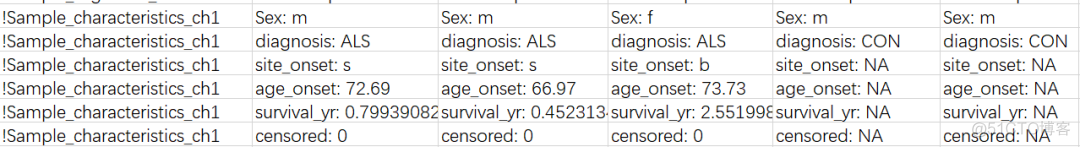
如果GEO里面找不到,可以去查看数据对应的文献或者联系文章作者来获取。

·end·

一个只分享干货的
生信公众号
