DiffBind是一个用于peak差异分析的R包,源代码保存在Bioconductor上,链接如下
http://www.bioconductor.org/packages/release/bioc/html/DiffBind.html
该R包采用了RNA_seq中差异基因表达的思路来进行peak的差异分析,和macs2的差异功能不同,DiffBind需要依赖已有的peak calling结果,将peak区域当做RNA_seq中的基因区域,然后对这些区域进行定量和差异分析,其核心的差异分析通过调用RNA_seq中常用的R包来实现,支持以下3种差异分析的R包
RNA_seq中进行定量,需要比对的bam文件和基因注释gtf文件, 类似地,DiffBind需要提供样本比对的bam文件以及peak calling得到的peak区域结果文件。为了方便导入,DiffBind提供了一个接口,将导入文件的相关信息保存在一个文件中,该文件内容示意如下
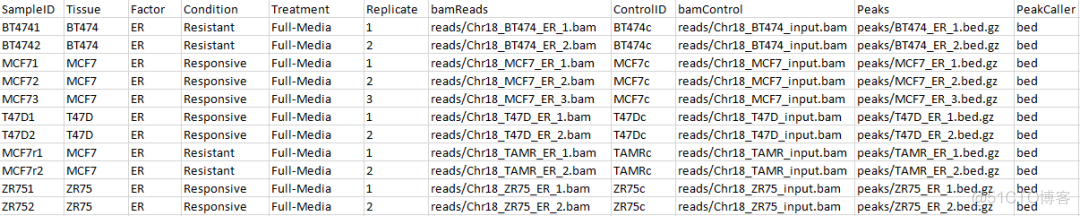
格式为csv, 这个表格的设计是为了考虑兼容性,最大可能的保留实验相关的所有信息。在实际分析中,可能有很多列没有对应信息,直接空值即可。值得注意的是,在ATAC中,样本没有对应的control, 这里control相关的信息为空就好,实际上这里的control也只是列在表格中,定量和差异分析时并不会用到control样本的数据。
Diffbind进行了高度封装,所有的函数都围绕一个自定义的DBA对象为中心,根据自己的数据整理好上述表格,然后通过以下几步代码就可以直接完成差异peak分析了
library(DiffBind)tamoxifen <- dba(sampleSheet="tamoxifen.csv")
tamoxifen <- dba.count(tamoxifen, summits=250)
tamoxifen <- dba.contrast(tamoxifen, categories=DBA_CONDITION)
tamoxifen <- dba.analyze(tamoxifen)
tamoxifen.DB <- dba.report(tamoxifen)
从函数名称也可以看出,从DBA对象开始,整个过程分为以下4步
需要注意的是,DiffBind要求必须有生物学重复,每组至少有两个样本,否则在contrast那一步会报错,源代码对对组内样本的个数进行了限制
if(minMembers < 2) {stop('minMembers must be at least 2. Use of replicates strongly advised.')
}
除了差异分析功能,DiffBind还提功了丰富的可视化功能,具体用法请参考官方文档。
·end·

一个只分享干货的
生信公众号
