基因组浏览器可以直观的展示基因组及其各种注释信息,是展示NGS数据和分析结果的利器。在维基百科中,将基因组浏览器定义为一种展示生物数据库中基因组学数据的图形化界面,可以用于展示和查看基因结构,蛋白,基因表达,调控,突变,比较基因组等多种信息的软件。
基因组浏览器有很多种,常见的几种列表如下
其中前三者都是基于网页的,而IGV则提供了独立的安装包,允许在本地运行。IGV由大名鼎鼎的Broad Institue团队开发和维护,官网如下
https://software.broadinstitute.org/software/igv/
该软件支持芯片数据,NGS数据,基因组注释等多种类型的数据,除了本地化安装外,也提供了web服务,网址如下
https://igv.org/app/
无需下载安装,只需联网,打开浏览器即可使用。本地化的软件采用java语言进行开发,下载地址如下
https://software.broadinstitute.org/software/igv/download
支持Windows, Mac, Linux多种平台,同时还提供了命令行版本的运行工具igvtools。软件的安装非常简单,直接下载对应平台的安装包即可。安装好之后,双击图标即可启动,界面如下
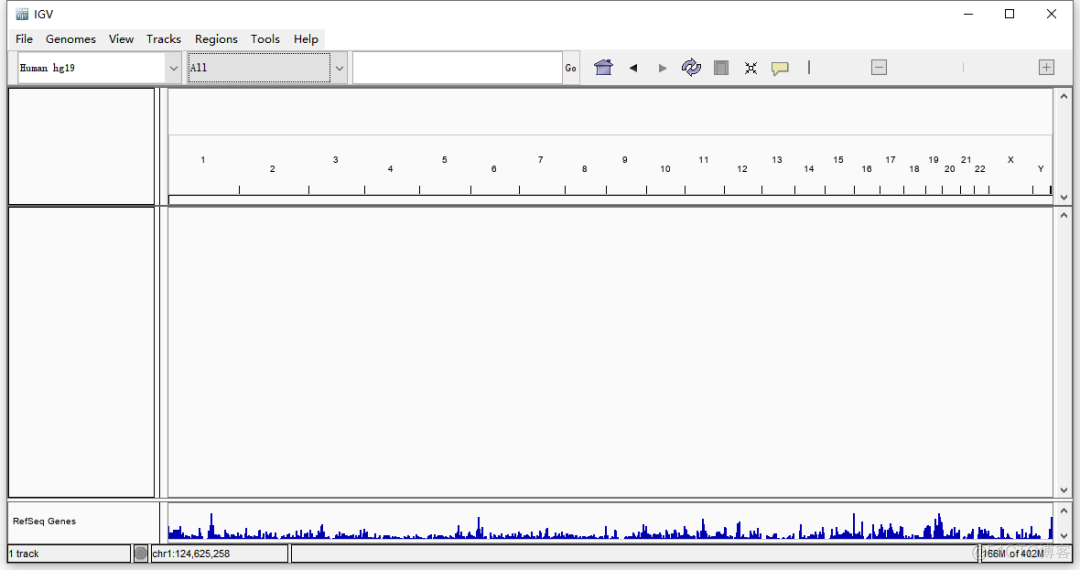
在IGV中,各种类型的信息以行为单位进行展示,每一行称之为track, 不同类型的数据其可视化方式不同。用IGV展示数据,基本步骤如下
1. 导入参考基因组
默认情况下,软件会导入hg19的参考基因组,启动时会看到如下所示的界面
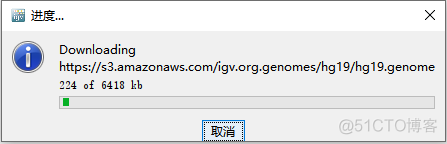
可以看到是从亚马逊的云服务器上下载了一个后缀为.genome的文件,该文件包含了基因组的序列和基因结构信息。导入之后,就可以按照染色体名称,染色体区域或者基因名称来快速查看感兴趣的基因组区域。
在这个服务器上,还提供了很多其他的基因组文件,通过点击菜单栏的Genomes->Load Genome From Server, 可以查看所有支持的物种,图示如下
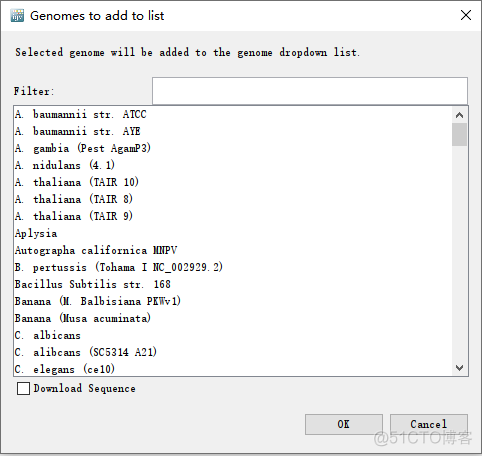
如果你的物种不在这个列表内,或者你想用自己的数据来构建参考基因组,也是可以的。利用基因组序列和基因结构信息,自己构建一个.genome的文件,然后导入到软件中即可,创建的方法后续再详细介绍。
2. 导入需要展示的数据
基因组导入之后,参考坐标系就有了。接下来就是导入需要展示的数据,IGV支持多种格式的数据,图示如下
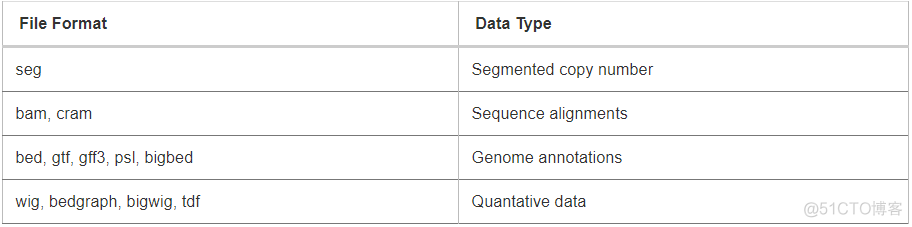
利用文件后缀来识别不同的格式,通过点击菜单栏的Files->Load From File按钮,即可导入。不同格式的文件展示形式不同,以tdf等定量结果为例,默认的展示结果是柱状图,示意如下

左侧为track name, 右侧为数据的柱状图展示,并且标记处了数据的范围,右键点击这个track, 可以设置tack name, track height, color等属性。
以上就是IGV的基本用法。IGV在chip_seq, m6AATAC等文章中使用广泛,可以很好的展示reads的分布情况,示例如下
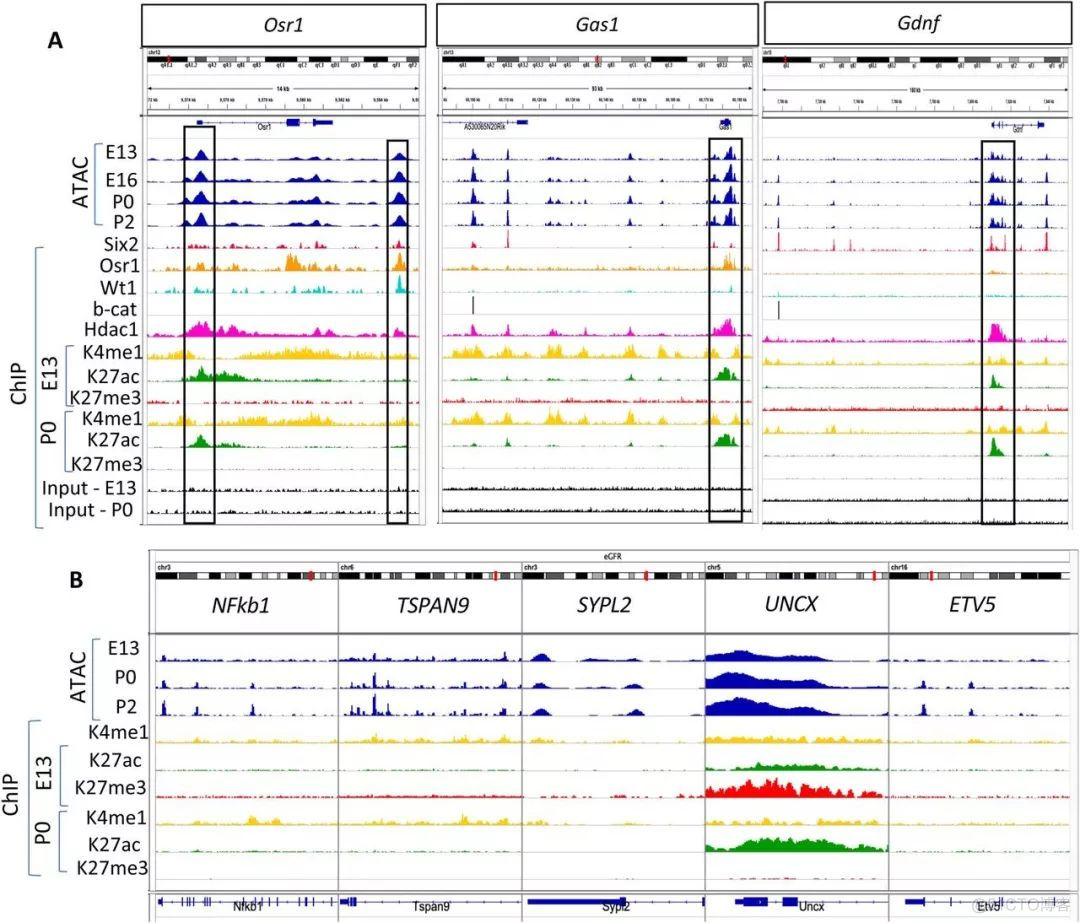
将IGV的原始输出结果用PS等工具进行排版和美化,就可以实现上图中的效果。
·end·

一个只分享干货的
生信公众号
