metasoft是一款对多个study的GWAS分析结果进行meta分析的工具,该软件执行速度快,而且配套了画森林图的工具,非常的方便,官网如下
http://genetics.cs.ucla.edu/meta/
在处理不同study的异质性时,支持以下4种模型
和metal类似,metasoft也只需要一个配置文件即可,配置文件的内容示意如下
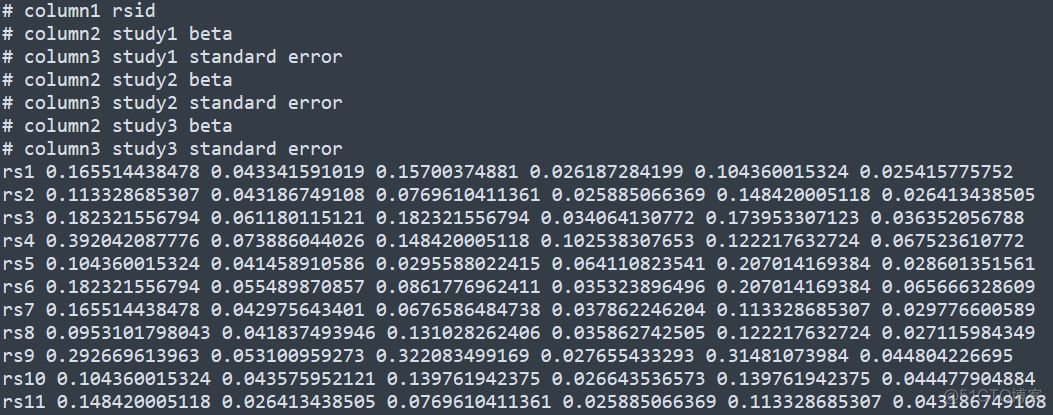
#开头的行是我后期加上去的,解释了每一列的含义,该文件以空格为分隔符,每一行为一个SNP位点,对于每个study的GWAS分析结果,以两列为单位进行展示,第一列为回归系数beta值,第二列为回归系数对应的标准误SE, 上述文件表示3个study的关联分析结果。
准备好输入文件之后,就可以运行该软件了,基本用法如下
java -jar Metasoft.jar -input example.txt默认输出文件名称为out, 其列数很多,每一列的含义解释如下
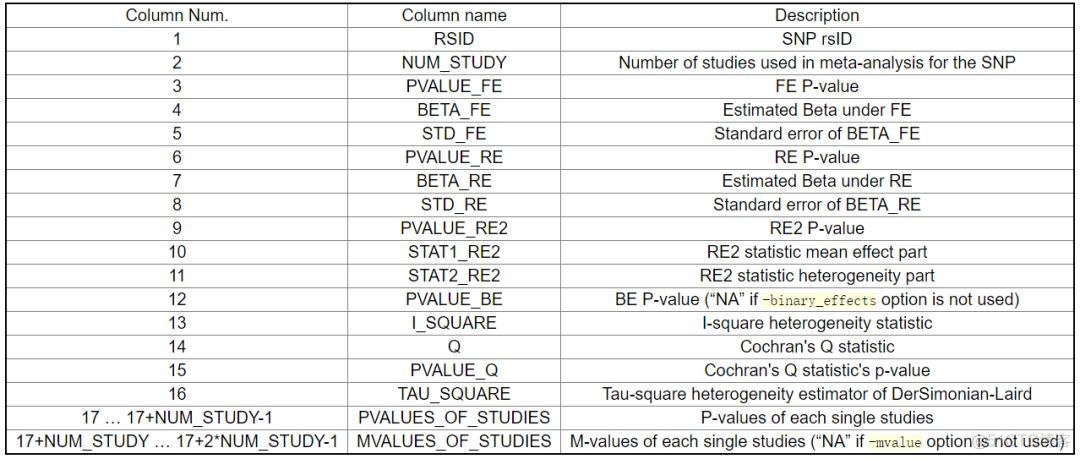
根据不同的effect 模型, 综合所有study的研究结果,给出一个总的beta值,标准误以及p值,以固定效应模型FE为例,对应的有以下3列
同时为了量化不同study之间的异质性,使用了Cochran’s Q检验,该检验的统计量称之为Q, 并在此基础上定义了新的统计量I square, 计算公式如下
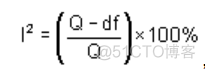
通常情况下,I square的值在0-0.25之间,认为是较低异质性;0.25-0.5之间,中等程度的异质性,0.5到0.75之间,真实存在异质性,0.75以上,异质性很大。
该工具还包含了一个名为ForestPMPlot的工具,可以基于metasoft的结果绘制森林图,基本用法如下
python pmplot.py \data/input.txt \
data/output.txt \
study.name.txt \
study.order.txt \
rs32075748 \
Apoa2 \
Apoa2.pdf
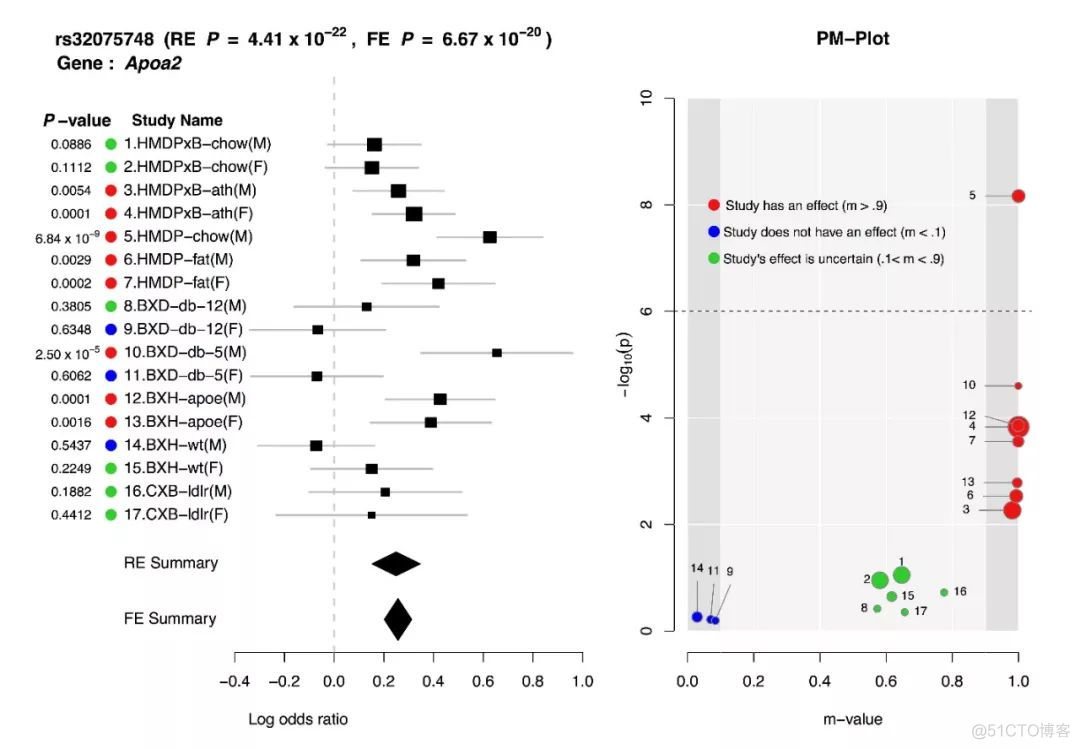
第一个参数为metasoft的输入文件,第二个参数为metasoft的输出文件,第三个参数为每个study的名称,第四个参数为每个study对应的顺序,第五个参数为需要绘图的SNP ID,第六个参数为对应的基因,第七个参数为输出图片的名称,图片示意如下
利用该软件,不仅可以进行gwas meta-analysis, 还可以快速绘制森林图,推荐大家使用。
·end·

生物信息入门
只差这一个
公众号
