作为最广泛使用的关联分析工具,plink支持卡方检验,费舍尔精确检验,逻辑回归,线性回归等多种分析方法,用法简单,运行速度快。使用plink进行case/control逻辑回归, 只需如下所示的一句代码
plink —bfile sample —logistic —allow-no-sex —ci 0.95 —out out
输出结果保存在out.assoc.logistic文件中,内容如下所示
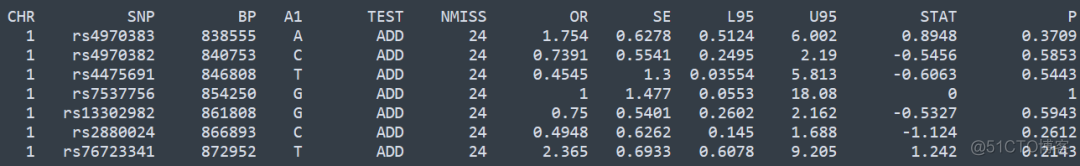
plink默认采用加性模型,针对单个SNP位点的基因型与case/control的分组进行逻辑回归分析,核心数据如下
genotype
AA
Aa
aa
Case
a
b
c
Control
d
e
f
以rs4970383为例,其对应的表格统计如下
genotype
AA
Aa
aa
Case
1
8
3
Control
2
3
7
对于这样的数据,用R语言进行逻辑回归分析,代码如下
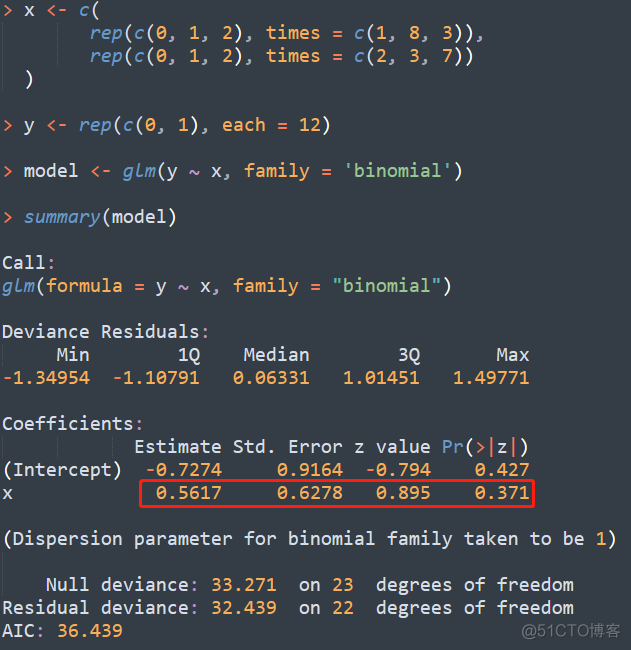
红框标记的部分就是逻辑回归的结果值,和plink的结果进行对比,可以看到,SE代表的就是标准误,STAT代表T检验的统计量,P代表T检验的p值。
默认显示OR值,也可以显示对应的回归系数值,代码如下
plink —bfile sample —logistic beta —allow-no-sex —ci 0.95 —out out
输出结果如下
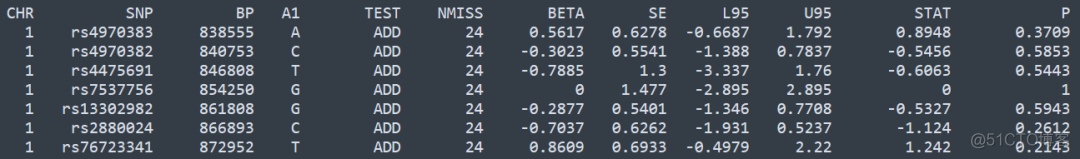
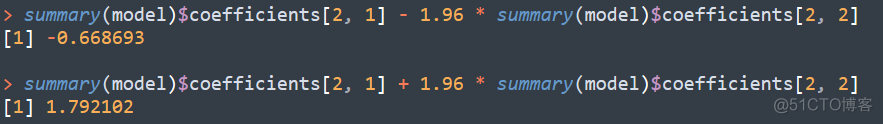
BETA列代表的就是回归系数了,而L95和U95对应的就是95%的置信区间了,在R中计算的结果如下
除了加性模型外,还支持显性,隐性模型,本质就是对基因型进行了划分,还是以rs4970383为例,显性模型中基因型统计如下
genotype
AA+Aa
aa
Case
9
3
Control
5
7
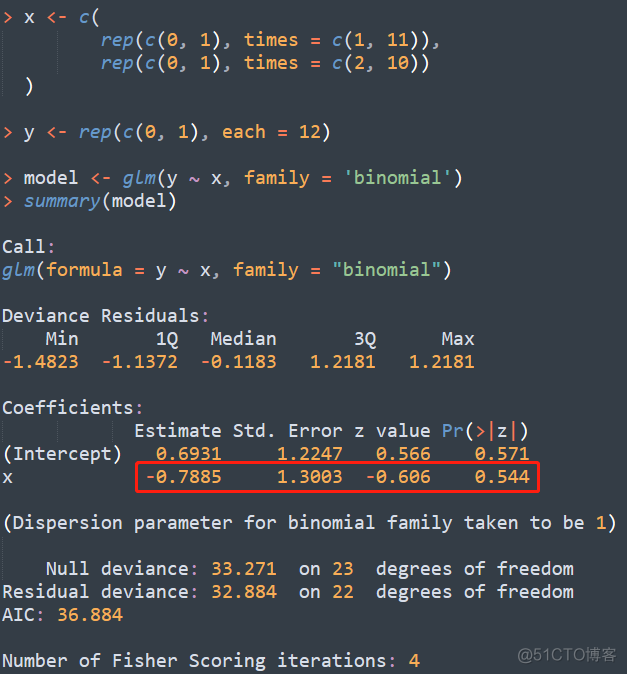
隐性模型中基因型统计如下
genotype
AA
Aa + aa
Case
1
11
Control
2
10
plink根据显性或者隐性模型进行逻辑回归的方法如下
plink —bfile sample —logistic dominant —allow-no-sex —ci 0.95 —out out
plink —bfile sample —logistic dominant —allow-no-sex —ci 0.95 —out out
分析结果如下

DOM代表显性模型,REC代表隐性模型,这里的OR值其实和逻辑回归没有太大关系,是直接根据上述2X2的表格计算出来的,根据OR值的公式,对于显性模型,OR值为
(9 7 ) / (3 5) = 4.2
隐性模型OR值为
(1 10) / (2 11) = 0.4545
用R来计算不同模型对应的p值,显性模型结果如下
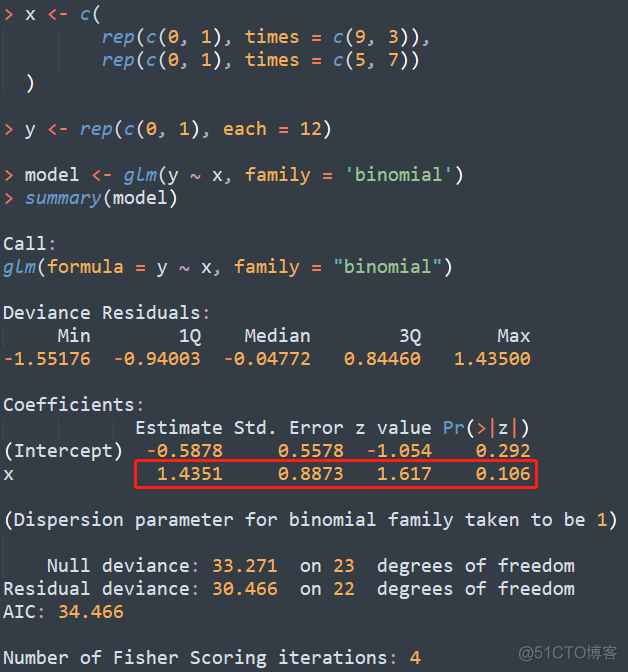
隐性模型结果如下
plink的优势之处在于同时对大量snp位点进行分析,如果只是对个别位点进行分析,自己用R语言也可以得到相同的结果。
·end·

