GWAS分析的结果通常使用曼哈顿图来进行展示,将所有的位点压缩到一张图上,好处是显而易见的,一眼可以看到染色体上所有位点的分布,其限制也很明显,分辨率不够高,对于感兴趣的染色体部分区域无法精细展示。
想要展示基因组的部分区域,比如某个基因上的关联分析结果,我们就需要用到locuszoom图。从名字也可以看出,这个图的本质就是对曼哈顿图中的部分基因组区域进行放大展示,可以提供更加详细的展示信息。
LocusZoom这个概念最早出现在以下文章中
https://academic.oup.com/bioinformatics/article/26/18/2336/208507
提供了一个在线网站,通过该网站可以轻松的展示基因组局部区域的关联分析结果,网址如下
http://locuszoom.org/
有两种图片的展示形式,一种生成静态的pdf文件,另外一种生成交互式的html页面,既可以上传自己的数据,也可以利用公开的数据集进行绘制,以静态图片为例,网址如下
http://locuszoom.org/genform.php?type=yourdata
输入文件内容示意如下
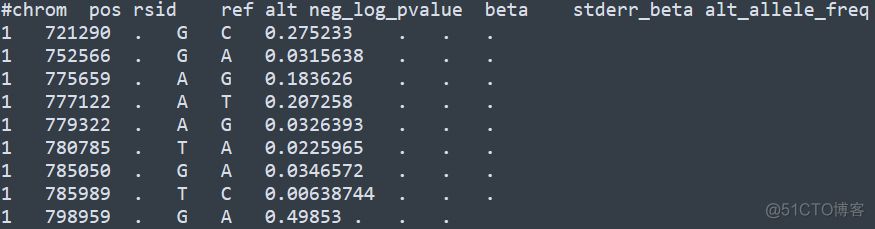
需要用到的基本信息包括chrom, pos, rsid, neg_log_pvalue,染色体位置需要和基因组版本对应,比如hg19/hg39, rsid表示SNP位点的rs号,有明确rs号的情况下可以指定rs 号来筛选绘图区域,neg_log_pvalue对应-log10pvalue。
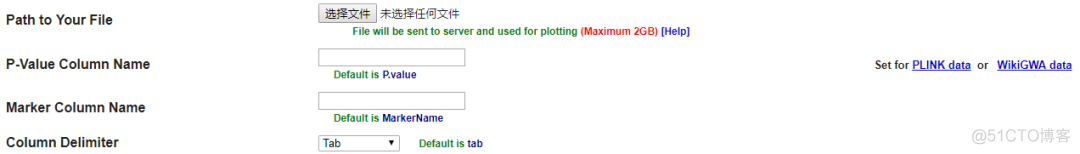
通过如下所示的对话框上传该文件,指定pvalue和rsid对应的列名,还有列的分隔符即可。
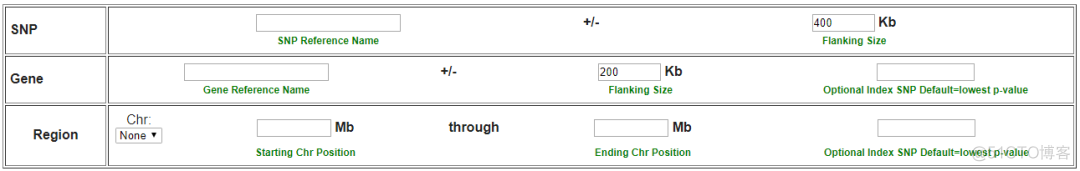
上传成功后,可以通过以下3种方式来指定绘图的区域
SNPGene
*Region
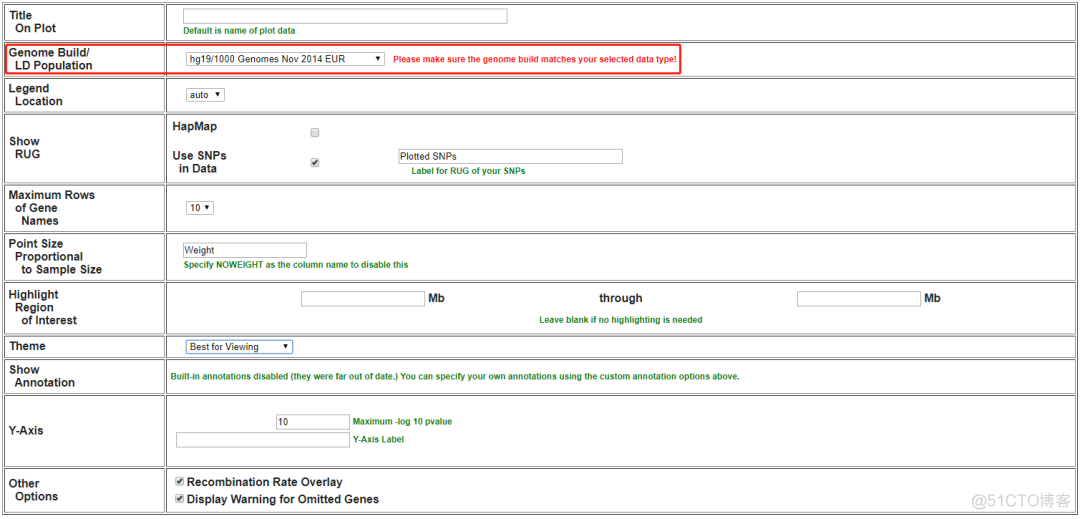
3种方式任选一种即可,除了pvalue信息外,locuszoom还会展示对应区域的基因注释以及连锁不平衡LD信息,这些信息保存在对应的数据库中,所以需要根据自己的数据选择对应的数据库版本
上述三个步骤到完成之后,点击plot data就可以出图了,图片内容示意如下
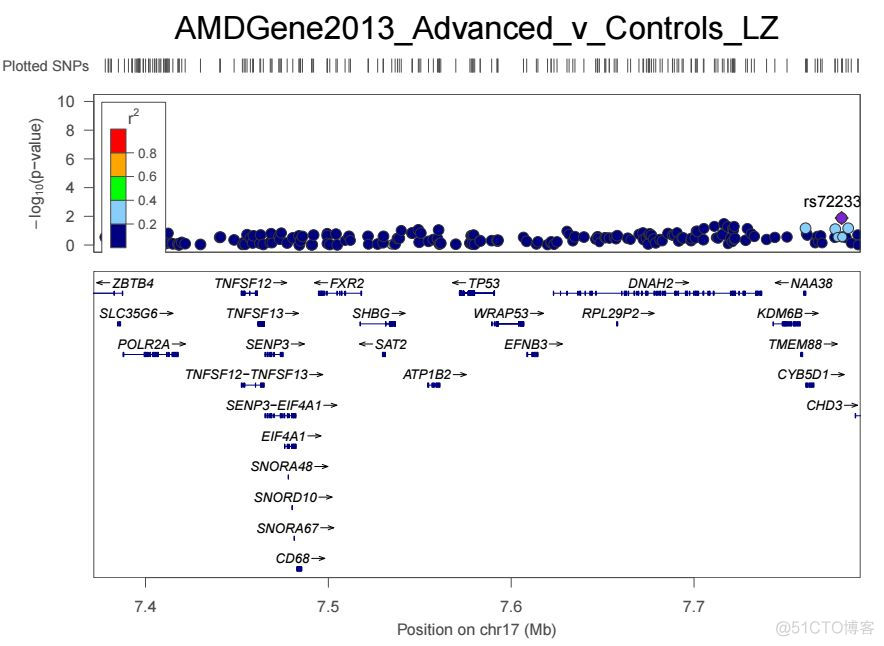
可以看到,locuszoom也没有什么神秘之处,就是在pvalue信息的基础上新增了基因注释和LD的信息。
该网站也提供了命令行版本,可以本地运行。通过LocusZoom图,可以精细化的展示特定基因组区域的关联分析结果。
·end·

生物信息入门
只差这一个
公众号
