IGV软件内置了很多的参考基因组,全部放在亚马逊的云服务器上,完整的参考基因组列表见如下链接 http://software.broadinstitute.org/software/igv/Genomes 每次打开IGV软件的时候,会从该
IGV软件内置了很多的参考基因组,全部放在亚马逊的云服务器上,完整的参考基因组列表见如下链接
http://software.broadinstitute.org/software/igv/Genomes
每次打开IGV软件的时候,会从该服务器下载一个默认的参考基因组,通常是hg19
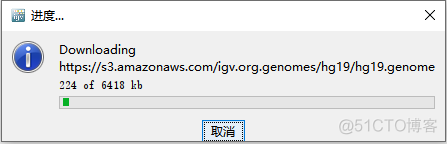
由于网络原因,可能会下载的非常慢,甚至是下载失败。对于这种情况,我们可以在本地构建一个参考基因组,每次从本地导入就好了,这样即使没有网络链接也可以正常使用。
构建一个本地的参考基因组,需要下列几种文件
上述4个文件中,只要基因组的fasta文件是必须的,其他3个文件都是可选的,通常情况下,只需要基因组序列和基因结构文件就可以满足需求了。需要注意的是,IGV不支持压缩文件,对于压缩文件,必须解压缩之后再使用。
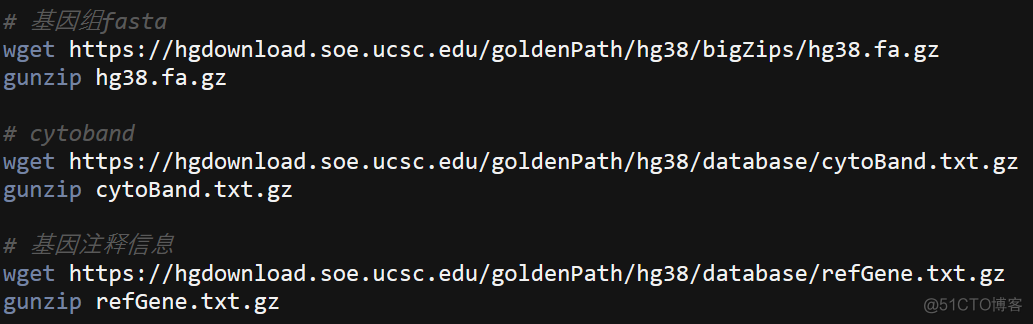
以hg38为例,展示下具体用法,首先从UCSC下载上述几种文件
对于fasta文件,要建立后缀为fai的索引,命令如下
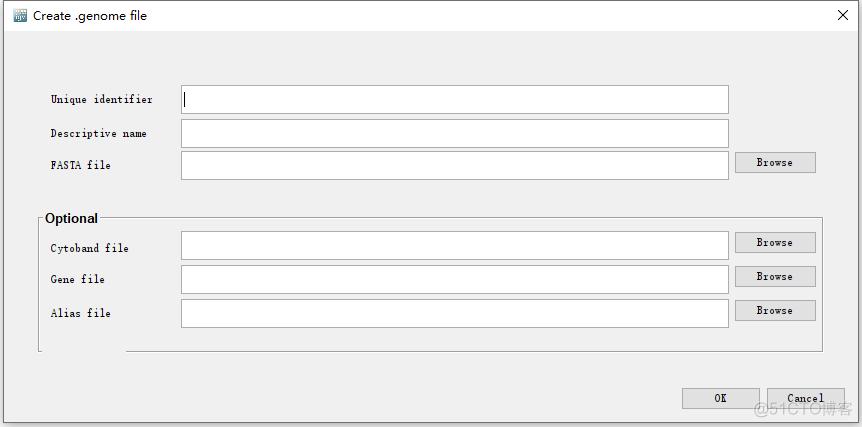
samtools faidx hg38.fa建好索引号,将上述几个文件放在同一个目录下,然后打开IGV, 点击菜单栏的Genomes->Create ,genome File,会弹出如下所示的输入框
选择对应的输入文件即可,示例如下
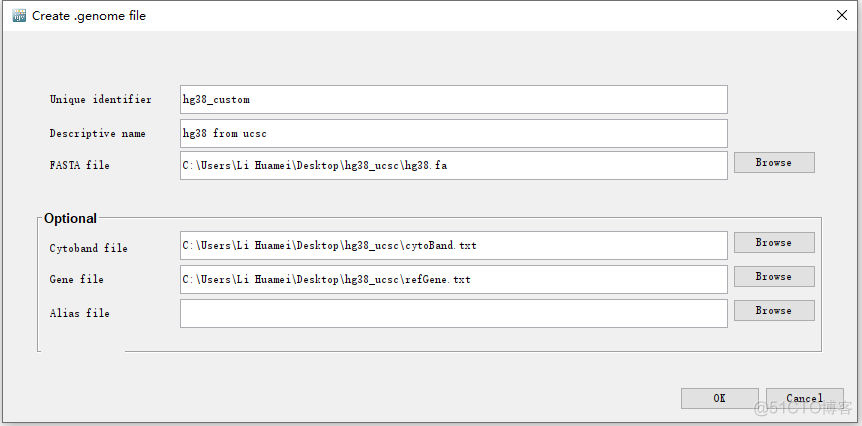
点击OK,选择一个目录来保存后缀为.genome的参考基因组即可。创建成功后,软件会自动导入该参考基因组,而且自定义的参考基因组名称也会出现在下拉列表里,下次使用时, 可以从下拉菜单选择本地的参考基因组。
通过创建本地化的参考基因组,可以避免网络差用不了IGV的情况,也大大扩展了IGV支持的物种列表。
·end·

一个只分享干货的
生信公众号
