对于基因组变异位点的存储,除了VCF外,还有一种常见的文件格式——MAF,是专门针对human突变位点注释信息的存储而定义的一种文件格式,最早在TCGA项目中广泛使用,在一个文件中同
对于基因组变异位点的存储,除了VCF外,还有一种常见的文件格式——MAF,是专门针对human突变位点注释信息的存储而定义的一种文件格式,最早在TCGA项目中广泛使用,在一个文件中同时包含所有样本的SNV和对应的注释信息,详细的格式介绍可以查看以下文章
- MAF:Mutation Annotation Format格式简介
本文的重点是如何使用IGV来查看MAF文件, 之所以说查看,是因为对于MAF的可视化,有更专业的工具
- maftools: 可视化maf文件的神器
IGV作为一款基因组浏览器,最大的特点是动态地查看基因组上各种信息的分布,对于MAF文件,导入IGV之后,首先会看到如下所示的柱状图
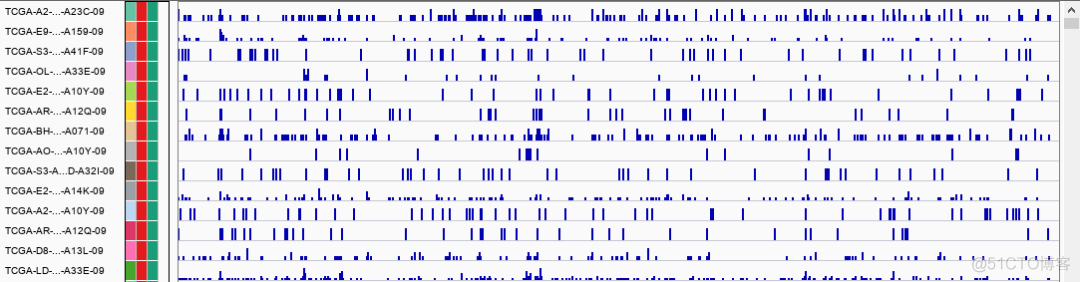
MAF文件中记录了多个样本的突变位点注释信息,上图中每一行表示一个样本,柱子的高低表示该区域突变位点的个数。进一步放大到单条染色体水平,可以看到如下所示的图
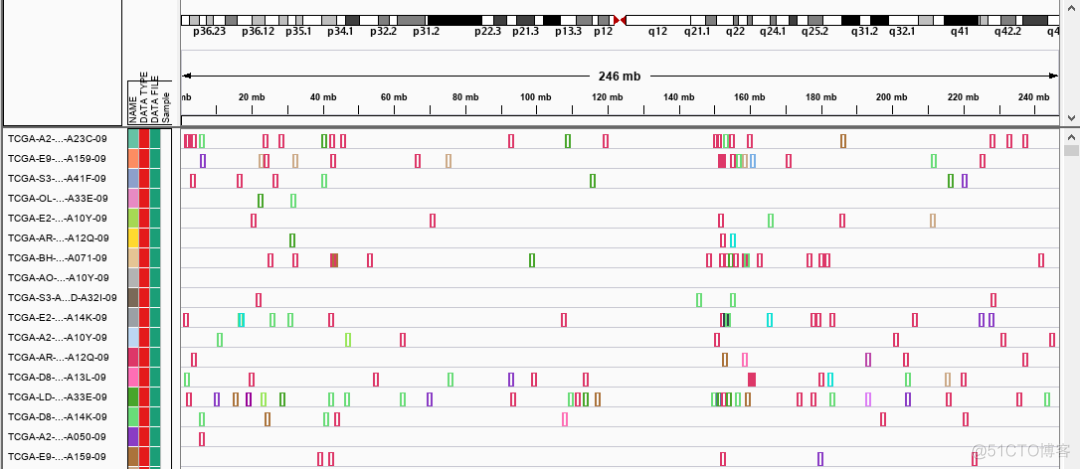
不同的突变类型用不同颜色的矩形表示,突变类型和颜色的对应关系可以通过菜单栏中的view->preferences中进行查看,示意如下
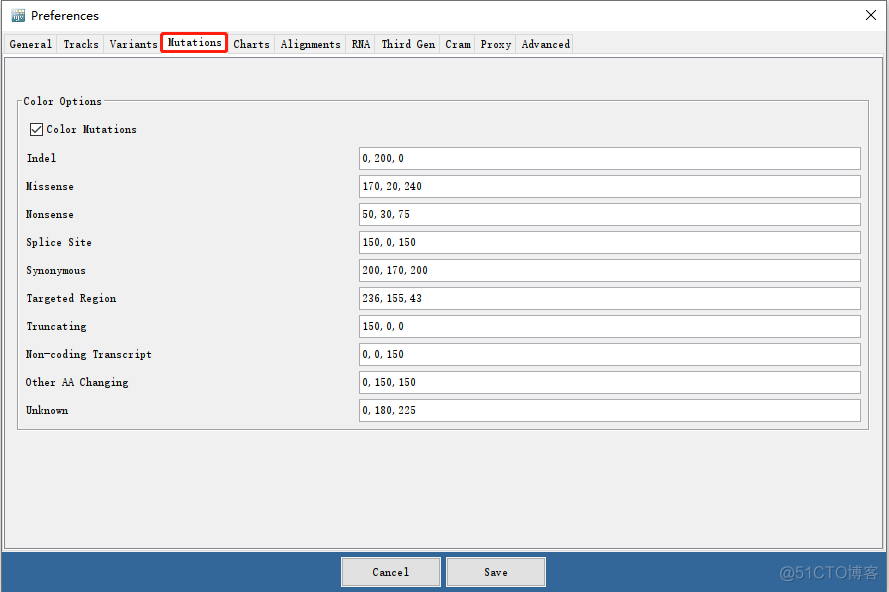
当然你也可以更改配置,将突变类型设置成你想要的颜色。对于MAF和VCF而言,IGV的长处都不是可视化,而是允许我们方便的查看任意基因组区域上突变位点的信息。
除了上述基本功能,IGV还支持将MAF中的突变位点与其他信息想叠加,详细的用法请查看以下链接
https://software.broadinstitute.org/software/igv/MutationData
·end·

一个只分享干货的
生信公众号
【本文来源:香港服务器租用 http://www.558idc.com/st.html欢迎留下您的宝贵建议】